Unser Service-Portfolio – Alle Konstellationen sind möglich
Was ist ein Exom?
Das Exom umfasst alle proteinkodierenden Regionen (Exons) der etwa 23.000 Gene im menschlichen Genom. Obwohl das Exom nur etwa 1 %-2 % des gesamten Genoms ausmacht, befinden sich etwa 89 % aller bekannten krankheitsverursachenden Varianten in Exons. Die Sequenzierung des gesamten Exoms ermöglicht die simultane Analyse von Genen in beliebiger Kombination. Werden, wie bei der Trio-Exom-Diagnostik, auch die Exome weiterer Familienmitglieder sequenziert, kann eine exomweite Segregationsanalyse durchgeführt werden. Im Vergleich zu Untersuchungen kleinerer Gen-Sets steigt so die Wahrscheinlichkeit, die genetische Ursache komplexer Phänotypen zu finden – und das in kürzerer Zeit.
Was ist eine Exom-Diagnostik?
Exom-Diagnostik ist der optimale genetische Test für Patientinnen und Patienten mit komplexen, heterogenen und unspezifischen Symptomen. Sie unterstützt Ärztinnen und Ärzte bei der Diagnosestellung, oft nachdem deren Patientinnen und Patienten Jahre der Ungewissheit erlebt haben.
Wir verfolgen das Ziel alle genetisch bedingten Krankheitsfälle zu lösen. Dafür haben wir einen innovativen diagnostischen Ansatz entwickelt, der deutlich über die Grenzen klassischer Exom-Diagnostik hinausgeht: ExomeFocus® und ExomeXtra® (Single & Trio) bieten eine effiziente Lösung für jedes Patientenszenario und jede Familienkonstellation.
Alle Erkenntnisse der Gendiagnostik vereint in unserer Exom-Diagnostik
Mit dem neuesten Update unserer ExomeXtra®-Anreicherung bieten wir Ihnen die innovativste Lösung in der Exom-Diagnostik. Entwickelt, um die umfassendsten Sequenzierdaten zu generieren, bietet unsere Exom-Diagnostik eine unübertroffene Grundlage für die beste verfügbare genetische Diagnostik mit herausragenden Eigenschaften.
Extra smartes klinisches Design
CeGaTs kundenspezifische Exom-Anreicherung ist das Ergebnis von mehr als 15 Jahren Optimierung von Anreicherungsdesigns für den klinischen Einsatz. Im Gegensatz zur Standard Exom-Sequenzierung (WES, whole exome sequencing) berücksichtigen CeGaTs Exom-Services nicht nur die proteinkodierenden Regionen, sondern alle bekannten krankheitsverursachenden Regionen im gesamten Genom. Mit mehr als 38.000 intronischen und intergenischen Varianten enthält CeGaTs hauseigene Exom-Diagnostik alle Bereiche des Genoms, die in der Human Gene Mutation Database (HGMD) und ClinVar, der größten öffentlichen Datenbank für Genotyp-Phänotyp-Beziehungen, als krankheitsrelevant beschrieben werden. Darüber hinaus wird das gesamte Genom in gleichmäßigen Abständen mit Sonden abgedeckt, um eine genomweite Array-ähnliche Identifikation von Kopienzahlveränderungen (copy number variants, CNVs) zu gewährleisten. Unsere Exom-Anreicherung umfasst auch eine klinisch relevante Auswahl von nicht-kodierenden RNA-Genen, flankierende intronische Sequenzen, kryptische Exons sowie das gesamte mitochondriale Genom.
Durch die kontinuierliche Verbesserung unseres Laborverfahrens haben wir eine sehr einheitliche und vollständige Abdeckung aller diagnostischen Zielregionen erreicht. Zur Verbesserung der Sensitivität und zur Erkennung von Mosaizismen sequenzieren wir ExomeFocus® und ExomeXtra® mit einer durchschnittlichen diagnostischen Abdeckung von > 100x.
Damit bildet CeGaT’s ExomeXtra® die ideale Grundlage für die umfassendste molekulargenetische Diagnostik.
Extra tiefgehende Analyse
CeGaTs Datenauswertung geht über die normale Exom-Diagnostik hinaus und erzielt höhere Aufklärungsraten. Wir berücksichtigen Kopienzahlveränderungen (copy number variants, CNVs), einschließlich komplexheterozygoter Kombinationen von Sequenzvarianten (single nucleotide variants (SNV), kleine Insertionen und Deletionen (small insertions and deletions, (Indels)) mit CNVs. Die Sequenzierdaten werden routinemäßg auf Expansionen von DNA-Repeats untersucht, die mit den erfassten Phänotypen in Zusammenhang stehen.
Es können unterschiedliche Anzahlen von Familienmitgliedern in die Analyse eingeschlossen werden, wie z. B. Duo (2) oder Trio (3). Bei Trio ExomeXtra® berücksichtigen wir Varianten in Genen mit reduzierter Penetranz, variabler Expressivität und sogar Imprinting-Effekte (IVERP). Bei der Trio-Analyse schließen wir zusätzlich die Erkennung von uniparentalen Hetero- und Isodisomien (UPD) ein.
Extra aufschlussreiche Ergebnisse
CeGaT kombiniert menschliches Know-how mit bioinformatischer Auswertung. Die hauseigene Software generiert Daten, die von mehreren wissenschaftlichen Expertinnen und Experten ausgewertet werden, um den bestmöglichen medizinischen Befund zu erstellen. CeGaTs interdisziplinäres, hochqualifiziertes Team nutzt stets die neueste Literatur zur Dateninterpretation. Komplexe Konstellationen werden mit Bioinformatikerinnen und Bioinformatikern diskutiert und anschließend von unseren Fachärztinnen und Fachärzten für Humangenetik überarbeitet, um die klinische Nutzbarkeit zu maximieren.
Smarter als Genom
Die Genom-Sequenzierung (WGS, whole genome sequencing) scheint die umfassendste verfügbare genomische Analyse zu sein. Im Vergleich zur Exom-Sequenzierung (WES) wird bei der WGS die Tiefe der Abdeckung (Empfindlichkeit) gegen die Reichweite (prozentualer Anteil des dargestellten Genoms) eingetauscht. Während WGS ein hervorragendes Instrument für die Forschung ist, erfordert der diagnostische Einsatz eine hohe Sensitivität, und der begrenzende Faktor ist die Interpretierbarkeit der entdeckten Varianten. Unsere Analyse zeigt, dass medizinisch relevante Regionen in einem gut konzipierten Exom besser repräsentiert sind.
CeGaTs ExomeXtra® erfasst mehr relevante Regionen mit höherer Abdeckung und bietet somit eine höhere Sensitivität als 30x WGS. Es berücksichtigt nicht nur die kodierende Region, sondern auch alle intronischen Varianten, von denen bekannt ist, dass sie mit einer Krankheit assoziiert sind, sowie die mtDNA und zusätzliche Zielsequenzen aus internen Datenbanken. Dies erhöht die Aufklärungsraten und ermöglicht es uns, Mosaizismus zu erkennen, welcher bei den meisten WGS-Analysen systematisch übersehen wird. Darüber hinaus bringt das integrierte Backbone über das gesamte Genom die CNV-Erkennung von CeGaTs ExomeXtra® auf das gleiche Niveau wie Array-CGH-Analysen.
WGS ist bereits ein hervorragendes Instrument für die Forschung. In der klinischen Praxis bietet ExomeXtra® die beste Kombination aus WES, WGS und Array-CGH und liefert damit die meisten genomischen Erkenntnisse zur Unterstützung der Patientenversorgung.
Tabelle 1: Vergleich zwischen Whole-Genome-Sequenzierung (WGS) und ExomeXtra®. Die Abdeckung bezieht sich auf die durchschnittliche klinisch nutzbare Abdeckung nach Alignment-Filterung (nur eindeutige Zuordnung, Entfernung von Duplikaten und überlappenden Read-Ends, etc.).
WGS | ExomeXtra® | |
Durchschnittlicher Abdeckungsgrad | ~ 30x | ~ 120x |
Total GB Sequenziert | 100 | 17,5 |
≥ 20x Abdeckung | ||
| 91,4 % | 98,3 % |
| 87,2 % | 98,3 % |
≥ 30x Abdeckung | ||
| 54,9 % | 98,1 % |
| 52,2 % | 97,1 % |
Ein detaillierter Überblick über unsere Exom-Diagnostik
Selbst entwickelte Exom-Anreicherung | ✓ | ✓ | ✓ | ✓ |
In-house Datenanalyse und Variantenbestimmung | ✓ | ✓ | ✓ | ✓ |
Gemeldete Varianten der ACMG Klassen | 3/4/5 | 3/4/5 | 3/4/5 | 4/5 |
Variantenklassifikation mit detaillierten Angaben zu ACMG-Kriterien und -Skala | ✓ | ✓ | ✓ | ✓ |
Manuelle Genauswahl für die Interpretation | ✗ | ✓ | ✓ | ✓ |
IVERP Varianten* | ✗ | ✗ | ✓ | ✓** |
VUS-Reevaluierung | ✗ | ✓ | ✓ | ✗ |
Kosten | € | €€ | €€€ | €€€ |
Lösungsrate |
*Berücksichtigung von Varianten in Genen mit Imprinting-Effekten, variabler Expressivität und reduzierter Penetranz.
** Pränataldiagnostik bei auffälligen Ultraschallbefunden.
Zusatzleistungen
ACMG-Gene
Das zusätzliche Analyse der ACMG-Gene ermöglicht den Nachweis relevanter pathogener Varianten außerhalb des Phänotyps in einer definierten Gruppe von Genen mit therapeutischer Relevanz.1 Bei allen ExomeXtra®-Analysen ist diese Option für die Indexpatientin bzw. den Indexpatienten kostenlos. Für ExomeFocus® ist das Screening der ACMG-Gene gegen einen Aufpreis erhältlich. Wenn Sie diese Option wählen, erstellen wir für jede Person einen separaten Befund, in dem wir die Ergebnisse innerhalb der ACMG-Gene angeben. Details zu den Genen und assoziierten Erkrankungen finden Sie hier.
Upgrade ExomeFocus® ExomeXtra® zu Trio ExomeXtra®
Trio ExomeXtra® ist der Test mit der größten Erfolgsaussicht, die genetische Ursache einer Krankheit zu identifizieren. In Fällen, in denen die behandelnde Ärztin oder der behandelnde Arzt mit einem Einzelanalyse-Exom beginnt und später auf unser Trio ExomeXtra® erweitern möchte, bieten wir dies zu einem reduzierten Preis an.
HLA-Typisierung
Mit Hilfe der HLA-Typisierung kann die Gewebekompatibilität für allogene Transplantate, das individuelle Ansprechen auf Medikamente (PGX) sowie die Differentialdiagnose bestimmt werden. Die HLA-Typisierung kann auch zur Beurteilung der Krankheitsanfälligkeit verwendet werden (z. B. HLA-B27: Entzündungs- und Autoimmunerkrankungen). Sie erhalten einen zusätzlichen Befund, in dem die HLA-Allele angegeben sind.
Pharmakogenetik
Die pharmakogenetische Analyse ermittelt genetische Veränderungen, die die Wirksamkeit von Medikamenten beeinflussen. Betreffen genetische Varianten Proteine, die für die Verstoffwechslung von Substanzen zuständig sind, kann deren Verträglichkeit und Wirksamkeit stark verändert sein. Zu diesen Arzneistoffen zählen unter anderem Antidepressiva, Schmerzmittel, Neuroleptika, Chemotherapeutika, AIDS-Medikamente, Thrombosemedikamente, Anästhetika, Betablocker oder Statine.
Die verringerte Aktivität eines spezifischen Enzyms kann bei der Standarddosierung zu einem erhöhten Medikamentenspiegel führen, der nicht selten mit unerwünschten Nebenwirkungen einhergeht. Bei Medikamenten, die erst durch die Verstoffwechslung aktiviert werden, kann der therapeutische Effekt ganz ausbleiben. Ebenso führt eine erhöhte Enzymaktivität, aufgrund der daraus resultierenden erhöhten Abbaugeschwindigkeit des Arzneistoffes, zu einer unzureichenden Wirksamkeit der Therapie.
Bei der Pharmakogenetik-Option werden bekannte Varianten in 22 Genen analysiert, die an der Verstoffwechselung von Arzneimitteln beteiligt sind. Bei Vorkommen bestimmter Genvarianten kann die behandelnde Ärztin oder der behandelnde Arzt die Therapie individuell anpassen. Mithilfe der pharmakogenetischen Analyse können gravierende Nebenwirkungen minimiert sowie ein Versagen der Therapie vermieden werden.
Das “Xtra” unserer Exom-Diagnostik
Kopienzahlveränderungen (CNVs)
Neben Einzelnukleotid-Varianten (SNVs) und kleinen Insertionen und Deletionen (Indels) können auch Kopienzahlveränderungen (CNVs) für einen Phänotyp ursächlich sein. Genetische Tests, bei denen Deletionen und Duplikationen vernachlässigt werden, sind unvollständig und können zu falschen medizinischen Berichten führen. All unsere Exomanalysen beinhalten automatisch ein Deletions-/Duplikationsanalyse – ohne zusätzliche Gebühren. Unsere CNV-Analyse in Kombination mit unserer maßgeschneiderten Anreicherung erhöht die diagnostische Ausbeute.
Die CNV-Analyse erlaubt es uns, einzelne Exon-Deletionen mit einer Sensitivität von >81 % zu erkennen. Größere Deletionen von drei oder mehr Exons werden mit einer Sensitivität von mehr als >96 % nachgewiesen. Zusätzlich validieren wir unsere Analyse mit MLPA oder qPCR, sobald wir Deletionen oder Duplikationen finden, die mit dem Phänotyp der Patientin oder des Patienten assoziiert sind.
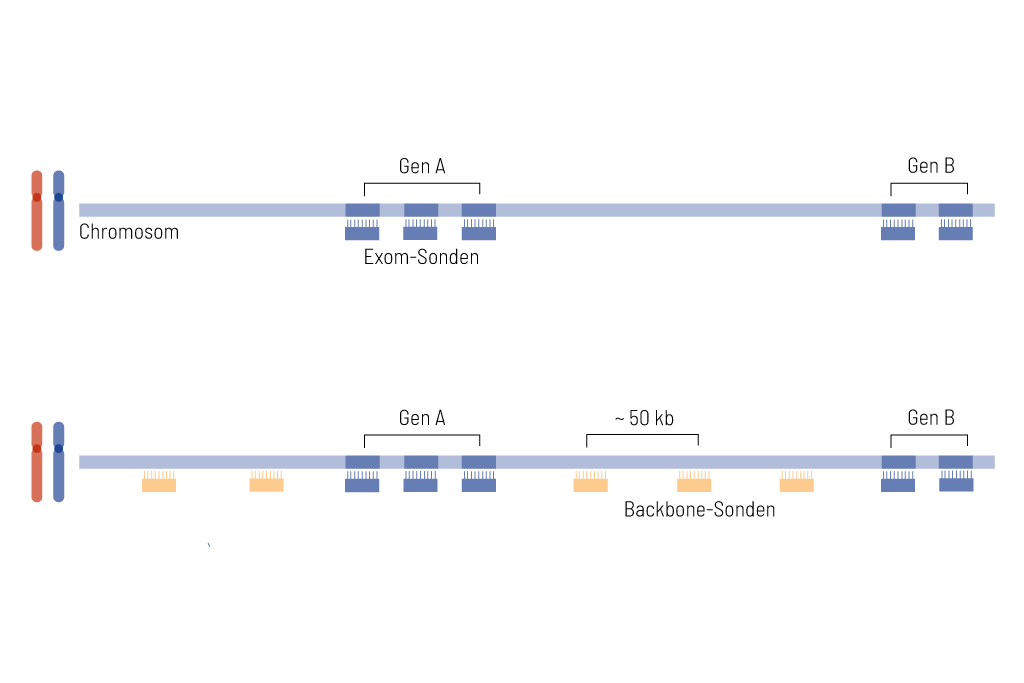
Außerhalb der kodierenden Exons deckt das CNV-Calling das gesamte Genom mit einer Array-ähnlichen Auflösung von ∼50 kb ab (Abbildung 1). Dies ermöglicht eine genaue Charakterisierung und Klassifizierung von klinisch relevanten CNVs und erlaubt die Erkennung von Bruchstellen in der intronischen Region. Um die korrekte Annotation dieser Strukturvarianten zu ermöglichen, ist die genomweite CNV-Analyse ein wesentlicher Bestandteil unseres Exom-Designs. Relevante Ereignisse werden in die medizinische Auswertung einbezogen. Darüber hinaus bestimmen und beschreiben wir stets die Qualität der CNV-Analyse in unserem medizinischen Bericht. Wir empfehlen den Versand von frischem EDTA-Blut, um die höchste Sensitivität des CNV-Calling zu erreichen.
Kombinierte Interpretation
CeGaTs Analysestrategie ermöglicht die Bestimmung von CNV/SNV-Kombinationen, um noch detailliertere und wichtigere Informationen zu erhalten. sind definiert als Vorkommen von sowohl einer SNV als auch einer CNV in heterozygotem Zustand, wobei mindestens eine der beiden der ACMG-Klasse 4 oder 5 (wahrscheinlich pathogen/pathogen) angehört.
Untersuchung von Repeat-Expansionen
Um die größtmöglichen Erkenntnisse aus unserem Exom-Datensatz zu gewinnen, suchen wir auch nach Expansionen von DNA-Repeats in mehreren Genen: SCA1 (ATXN1), SCA2 (ATXN2), SCA43 (ATXN3), SCA6 (CACNA1A), SCA7 (ATXN7), SCA17 (TBP), FXN, AR, CSTB, FMR1, HTT, JPH3, PABPN1. Wenn die Ergebnisse auf eine mögliche Repeat-Expansion hinweisen, die zum Phänotyp der Patientin oder des Patienten passt, bieten wir die Überprüfung mit einer unabhängigen Methode an.
Analyse des mitochondrialen Genoms
Das mitochondriale Genom ist in unserer Anreicherung enthalten und wird immer sequenziert. Varianten im mitochondrialen Genom werden ausgewertet, wenn der Phänotyp auf die Möglichkeit einer mitochondrialen Erkrankung hinweist. Bei der Pränataldiagnostik ist die Analyse der mtDNA immer inbegriffen.
VUS-Reevaluierung
CeGaTs VUS-Reevaluierung bewertet zuvor berichtete Varianten mit unklarer klinischer Signifikanz (VUS) neu, sobald neue wissenschaftliche Erkenntnisse zur Pathogenität der Variante vorliegen. Dies erhöht die Lösungsrate von CeGaT-Analysen.
Die Forschung im Bereich der Genetik macht große Fortschritte und erweitert kontinuierlich unser Wissen über krankheitsverursachende Varianten. Es ist daher wahrscheinlich, dass eine VUS mit der Zeit besser verstanden und als pathogen (wahrscheinlich pathogen/pathogen) oder gutartig (wahrscheinlich gutartig/gutartig) klassifiziert wird.
Im Falle einer solchen Re-Klassifizierung informiert CeGaT die behandelnden Ärztinnen und Ärzte. Die reklassifizierte Variante wird von uns im Hinblick auf das Krankheitsbild der Patientin oder des Patienten erneut interpretiert. Sollte die Variante im Zusammenhang mit dem Phänotyp eingruppiert werden, wird ein neuer medizinischer Befund erstellt. Dieser Service ist für Sie kostenlos (ExomeXtra®). Die VUS-Reevaluierung ist für die Betreuung und Behandlung von Betroffenen und deren Familienmitgliedern sehr relevant.
Fallbeispiel
Patient: Junge, 11 Monate
Klinische Symptome: Benigne Epilepsie mit tonisch-klonischen Anfällen
Primärbericht (November 2019): SCN8A c.802A>T (het.) VUS
VUS-Reevaluierung:
- ausgelöst durch die HGMD-Aktualisierung im Januar 2021
- basierend auf der Publikation Medlin et al., 20202
➔ Variante kann nun als “wahrscheinlich pathogen” eingestuft werden. Ein neuer Befund wurde zur Verfügung gestellt.
Referenzen
1 Gemäß den aktuellen Richtlinien des American College of Medical Genetics and Genomics; Einzelheiten zu Genen und damit verbundenen Krankheiten finden Sie unter www.cegat.de/acmg-gene.
2iMedlin, L. C., Bello-Espinosa, L. & MacAllister, W. S. Neuropsychological profiles of two patients with differing SCN8A-pathogenic variants. Applied neuropsychology. Child 11, 561–566; 10.1080/21622965.2020.1807983 (2022).
Unsere Akkreditierungen
Verbindliche Standards gewährleisten die Qualität unserer Arbeit: Unsere Laborleistung ist nach CAP/CLIA und DIN EN ISO 15189 akkreditiert.
Weitere Akkreditierungen und Zertifizierungen finden Sie hier.
Schauen Sie sich auch gerne folgende Bereiche an
Kontaktieren Sie uns
Sie haben noch eine Frage oder Interesse an unserem Service?
Diagnostik-Support
Wir unterstützen Sie auf Wunsch bei der Auswahl der diagnostischen Strategie – für jede einzelne Patientin und jeden einzelnen Patienten.