Die Analyse aus Liquid-Biopsy-Proben weist zellfreie DNA (cfDNA) nach, die von nekrotischen und apoptotischen Zellen in den Blutkreislauf abgegeben wird und ist somit eine optimale Alternative, wenn kein Tumorgewebe zur Verfügung steht oder zum Monitoring des Krankheitsverlaufes. Allerdings stammt nur ein Bruchteil der zirkulierenden DNA aus dem Tumor selbst. Daher sind hochsensitive Methoden erforderlich, um diese minimalen cfDNA-Konzentrationen nachzuweisen.
Das von uns entwickelte CancerDetect®-Panel basiert auf einer duplex UMI-basierten Technologie und weist die am häufigsten vorkommenden, therapierelevanten Treibermutationen nach. Darüber hinaus ist die Probenahme nicht invasiv und leicht wiederholbar, was CancerDetect® zu einem ausgezeichneten Ansatz für Monitoringanalysen macht. Aufgrund des hochsensitiven Nachweises tumorspezifischer Biomarker kann die Analyse der cfDNA optimal als Surrogatmarker für das Ansprechen der Behandlung im Rahmen der medizinischen Nachsorge eingesetzt werden.
Bei vielen somatischen Analysen, die auf Liquid Biopsy basieren, ist es hilfreich, die Veränderungen der Ergebnisse im Laufe der Zeit zu beobachten und sie im klinischen Kontext zu interpretieren. Mit dem neuen Layout des Monitoringbefundes werden die Daten von seriellen CancerDetect®-Analysen in einem Befund zusammengefasst und können auf einen Blick interpretiert werden. Die Ergebnisse der Sequenzierung jeder weiteren Probenentnahme werden in die bestehenden Befunde integriert. Anhand einer sich aktualisierenden Verlaufsgrafik und entsprechender Tabellen kann klar ein Trend relevanter Veränderungen anhand der Variantenfrequenz abgelesen werden. So können Aussagen über den Krankheitsverlauf getroffen, ein Therapieerfolg abgelesen oder das Auftreten von Resistenzmutationen rechtzeitig erkannt werden.
Sie sind in Deutschland versichert? Unsere Kolleginnen und Kollegen vom Zentrum für Humangenetik Tübingen beraten Sie gerne!
Die optimale Lösung für Monitoring und Nachsorge
Unser Versprechen an Sie
Service Details
- Die Liquid-Biopsy-Analyse ermöglicht den Nachweis von Varianten mit potenzieller therapeutischer Relevanz bei Patientinnen und Patienten, bei denen der Tumor nicht zugänglich ist. Damit können Informationen über den Tumor gewonnen und eine Behandlung ermöglicht werden (siehe Beispielbefund „Single/First Sample“). — Mehr erfahren
- Beinhaltet eine Liste aller in Frage kommenden Medikamente mit EMA und/oder FDA-Zulassung, für deren Anwendungsoption entsprechende Biomarker im Tumor nachgewiesen werden konnten (siehe Beispielbefund „Single/First Sample“) — mehr erfahren
- Hochsensitiver und präziser Nachweis der am häufigsten vorkommenden Treibermutationen in 31 Genen sowie von Fusionen in 6 Genen mit sehr niedrigen Allelfrequenzen durch den Einsatz duplex UMI-basierter Technologie (NAF ≥ 0,25 %)
- Hohe Abdeckung von 50.000–100.000x in den Rohdaten
- Die einfach zu entnehmende, nicht-invasive und wiederholbare Probennahme bietet die besten Voraussetzungen für Folgeuntersuchungen.
- Durch den sehr empfindlichen Nachweis tumorspezifischer Biomarker kann die Analyse der cfDNA eingesetzt werden, um die Tumordynamik in Echtzeit zu verfolgen und wenn nötig einzugreifen oder die Behandlung anzupassen (z. B. bei erworbener Arzneimittelresistenz).
- Bei seriellen Probenentnahmen werden die Ergebnisse vorangegangener Analysen automatisch mitberücksichtigt und der Verlauf übersichtlich dargestellt. Hier liegt der Fokus auf der möglichen Veränderung der Variantenfrequenzen wichtiger Treibermutationen über die Zeit. (siehe Beispielbefund „Monitoring“).
- Das System ist flexibel aufgebaut und betrachtet die Frequenzveränderung jeder somatischen Variante separat. Daher ist die Integration erstmals neu auftretender Veränderungen in den Verlaufsbefund jederzeit möglich.
Alle Untersuchungsergebnisse werden von uns in einem klar strukturierten und übersichtlichen medizinischen Befund mitgeteilt. Dieser beinhaltet eine Liste mit allen identifizierten klinisch relevanten Varianten und sich daraus ggf. ableitenden Therapieansätzen für jedes erste Sample einer Serie sowie für Einzelanalysen mit nur einem untersuchten Probenentnahmezeitpunkt.
Jeder medizinische Befund wird von unserem interdisziplinären Team bestehend aus Wissenschaftlerinnen und Wissenschaftlern, sowie Ärztinnen und Ärzten erstellt und besprochen. Damit garantieren wir Ihnen die höchste Qualität.
Beispielbefund „Single/First Sample“
Beispielbefund „Monitoring“
Unsere Standaranforderungen für Proben
Liquid Biopsy
Bei Liqui-Biopsy-Proben handelt es sich um Probenmaterial, dessen Abnahme nur mittels spezieller, die zellfreie DNA stabilisierender Entnahmeröhrchen erfolgen kann. Sollten Sie eine Untersuchung auf Basis von cfDNA planen, so verwenden Sie bitte derartige Röhrchen. Wir stellen diese gern zur Verfügung, nehmen Sie bitte hierfür rechtzeitig mit uns Kontakt auf.
- 3x 10 ml cfDNA-Röhrchen für Liquid Biopsy (bei Blut als empfohlener Probenart)
Falls Liquor oder Flüssigkeit aus Zysten als Ausgangsmaterial verwendet werden sollen, bitte Entnahmemenge entsprechend verringern falls nötig.
Geeignete Probentypen für die cf-DNA Isolation
- Blut
- Ascites
- Liquor
- Pankreas-Zysten-Flüssigkeit
Sprechen Sie uns gern an, wenn Sie andere Probentypen analysieren möchten.
Informationen zum Probenversand
Hier finden Sie weitere Informationen zum sicheren Versand Ihrer Probe.
Das macht unseren CancerDetect® Service besonders
Varianten mit potenzieller therapeutischer Relevanz
Wegweiser für potenziell wirksame Medikamente
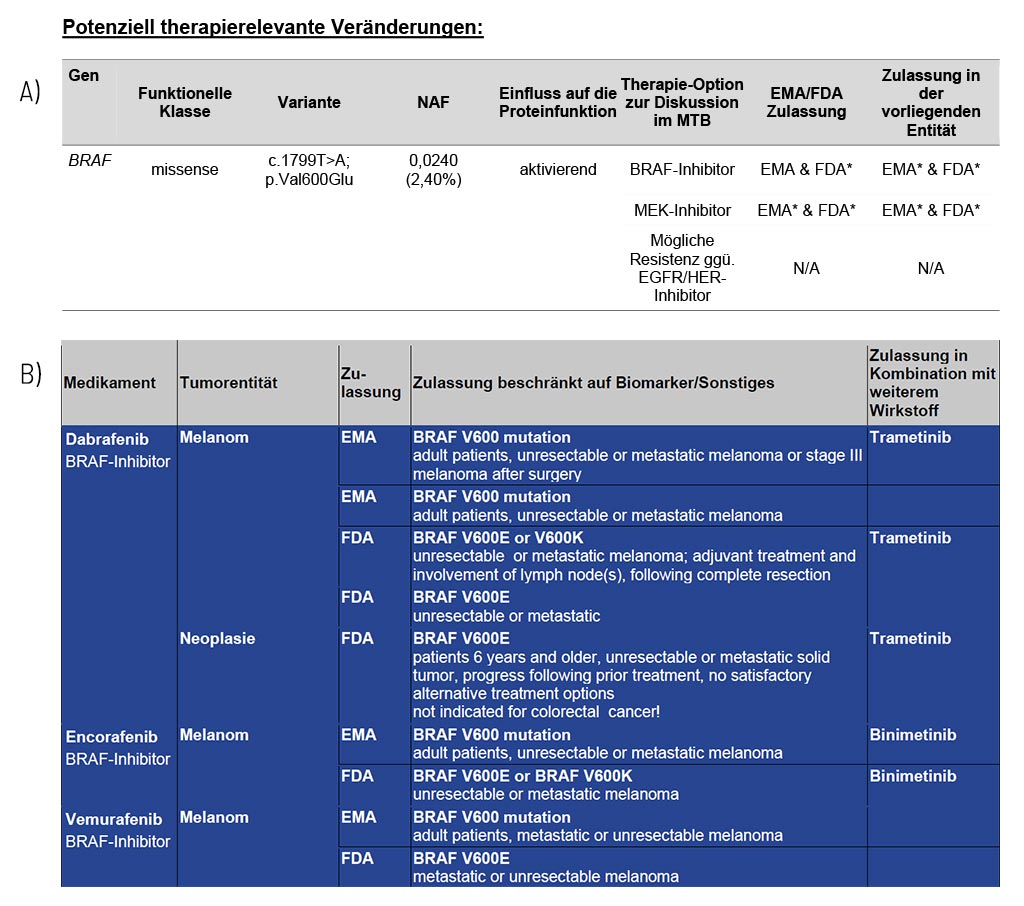
Die einzelnen therapierelevanten Veränderungen werden für jedes Gen detailliert dargestellt und die sich daraus ergebenden therapeutischen Optionen, einschließlich der EMA/FDA-Zulassung, angegeben (A). Diese Optionen bilden die Grundlage für die Diskussion in einem molekularen Tumorboard (MTB).
Im Anhang des medizinischen Befunds stellen wir eine umfangreiche Liste möglicher therapeutischer Strategien für jede identifizierte somatische Veränderung zur Verfügung (B). Diese Liste enthält neben den Medikamentenklassen und -namen auch deren Zulassung (FDA/EMA) und einschränkende Bedingungen. Die Übersicht über die therapeutischen Optionen wird in jedem ersten Befund einer seriellen Verlaufsdiagnostik in vollem Umfang ausgewiesen.
Beispielbefund: Beispiel für die entdeckte BRAF-Variante und die daraus resultierenden therapeutischen Optionen bei einer Patientin mit Melanom. Oberer Teil A): Ein Auszug aus Tabelle 1 des Befundes, der Varianten mit therapeutischer Relevanz auflistet. Unterer Teil (B): Ein Auszug der Medikamenten-Auflistung. Neben den abgebildeten Medikamenten werden auch andere Medikamente beschrieben.
CancerDetect® Anwendungen bei Monitoring
Für serielle Probenentnahmen werden ab der zweiten Probe automatisch Verlaufsbefunde erstellt, die eine beliebige Anzahl vorangegangener Analysen aus Liquid-Biopsy-Proben integrieren können. Der Verlaufsbefund erlaubt so dank seiner übersichtlichen Abbildungen und Tabellen mit einem Blick Veränderungen im Auftreten überwachter Tumormutationen zu erfassen. Hinweise auf ein Rezidiv können so frühzeitig entdeckt, Patientinnen und Patienten mit einer „stable disease“ regelmäßig einfach überwacht oder das Auftreten von Resistenzmutationen unter Therapie sofort im Therapieplan berücksichtigt werden.
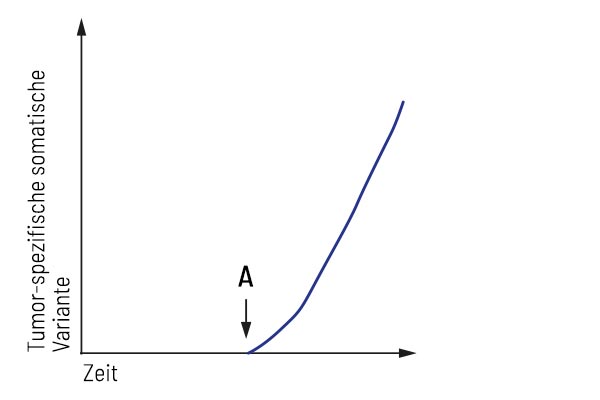
Anwendung „Rezidiv-Früherkennung“:
Nach der Behandlung wurde der Patient als tumorfrei betrachtet. Regelmäßige Liquid-Biopsy-Analysen ergaben ein Fortschreiten des Tumors, welches mit der Zunahme einer tumorspezifischen Variante und später auch mit dem klinischen Wiederauftreten des Tumors einherging. Der hochempfindliche Nachweis des Biomarkers kann ein Tumorrezidiv früher erkennen als herkömmliche bildgebende Verfahren.
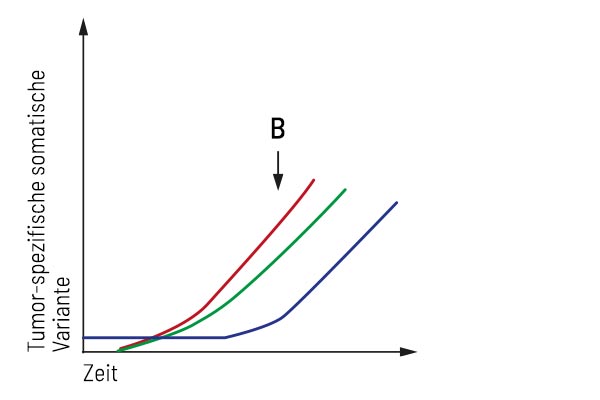
Anwendung „Monitoring während der Behandlung“:
Nach der Behandlung kam es bei dem Patienten zu einem schnellen Regress, der zu einem stabilen Zustand führte. Das longitudinale Monitoring liefert ein aktuelles molekulares Profil des Tumors und erkennt aufkommende Therapieresistenz rechtzeitig. Hier treten neben der Primärtumor-Mutation zwei weitere Subklone auf, die eine entsprechende Anpassung der Behandlung notwendig machen.
Fallbeispiel
Patientin und Indikation:
- 42 Jahre alt, weiblich, metastasierendes nicht-kleinzelliges Lungenkarzinom (Non-small cell lung cancer) mit einer EGFR-L858R-Mutation im Primarius
- Rezidiv nach initialem Ansprechen auf Afatinib-Behandlung
- Rezidiv inoperabel, keine Tumorbiopsie möglich
Primärer Befund:
Das Ergebnis einer Analyse der zellfreien DNA mit einem Standard-Panel für Lungenkrebs blieb negativ.
CancerDetect®-Befund:
Unsere CancerDetect®-Analyse ergab einen Tumorgehalt von 2 % in der Liquid Biopsy und wies die EGFR-L858R-Mutation, sowie zusätzlich eine EGFR-T790M-Mutation nach. Die EGFR-T790M-Mutation stellt einen der häufigsten Resistenzmechanismen gegen Tyrosinkinase-Inhibitoren (TKI) dar und tritt typischerweise bei NSCLC-Patientinnen und -Patienten nach Erstlinien-TKI-Behandlung auf. Bei dieser Patientin wurde die Behandlung mit dem EGFR-Inhibitor der dritten Generation Osimertinib angepasst.
Genverzeichnis
Es werden alle relevanten Varianten in einem beschriebenen Exon analysiert. Die Nummerierung der Exons bezieht sich auf die kodierenden Exons des jeweiligen Gens (CDS). Die Diagnose ist nicht auf die aufgeführten Beispiel-Hotspot-Mutationen beschränkt. Nicht beschriebene Exons und alle Varianten darin sind kein Bestandteil der Analyse.
Gene | NM_Nr. | Angereicherte Region (inkl. Beispiel Hotspot (HS)- Varianten) |
AKT1 | NM_005163.2 | Exon 2 (inkl. HS E17) |
ALK | NM_004304.5 | Exons 22-25 (inkl. HS F1174 , G1202, F1245, |
AR | NM_000044.6 | Exons 4, 5, 8 |
BRAF | NM_004333.6 | Exons 11, 15 (inkl. HS V600) |
CDKN2A | NM_000077.5 | Gesamte kodierende Region |
CTNNB1 | NM_001904.4 | Exons 2, 6, 7 (inkl. HS S37, S45, K335, N387) |
EGFR | NM_005228.5 | Exons 2, 3, 6, 7, 15, 18-21 |
ERBB2 | NM_004448.4 | Exons 8, 17, 19-21 (inkl. HS S310, R678, V842) |
ERBB3 | NM_001982.4 | Exons 3, 7-9, 23 (inkl. HS V104, E928) |
ESR1 | NM_000125.4 | Exons 4, 5, 7, 8 (inkl. HS K303, Y537, D538, E380Q, |
FGFR1 | NM_023110.3 | Exons 11-13 (inkl. HS N577, K687) |
FGFR2 | NM_000141.5 | Exons 6, 8, 11-13 (inkl. HS S252, N549) |
FGFR3 | NM_000142.5 | Exons 6, 8, 13 (inkl. HS R248, S249, Y375) |
GNA11 | NM_002067.5 | Exons 4, 5 (inkl. HS R183, Q209) |
GNAQ | NM_002072.5 | Exons 2, 4, 5 (inkl. HS T96, R183, Q209) |
Gene | NM_Nr. | Angereicherte Region (inkl. Beispiel Hotspot (HS)- Varianten) |
GNAS | NM_000516.7 | Exon 8 (inkl. HS 201) |
H3-3A | NM_002107.7 | Exon 1 (inkl. HS K27, G34) |
HRAS | NM_005343.4 | Exons 1-3 (inkl. HS G12, Q61) |
IDH1 | NM_005896.4 | Exon 2 (inkl. HS R132) |
IDH2 | NM_002168.4 | Exon 4 (inkl. HS R140, R172) |
JAK2 | NM_004972.4 | Exon 12 (inkl. HS V617) |
KIT | NM_000222.3 | Exons 9, 11, 13, 14, 17 |
KRAS | NM_004985.5 | Exons 1-3 (inkl. HS G12, G13, Q61) |
MET | NM_001127500.3 | Exons 13, 15, 18 (inkl. HS L982_D1028del, T1010, |
NRAS | NM_002524.5 | Exons 1-3 (inkl. HS G12, Q61) |
PDGFRA | NM_006206.6 | Exons 11, 13, 17 (inkl. HS D842) |
PIK3CA | NM_006218.4 | Exons 1, 4, 7, 9, 13, 20 |
PTEN | NM_000314.8 | Gesamte kodierende Region |
RET | NM_020975.6 | Exons 10, 11, 13-16 (inkl. HS C634) |
TERT | NM_198253.3 | Promotor HS c.-124 (C228), c.-146 (C250) |
TP53 | NM_000546.6 | Gesamte kodierende Region |
DNA-basierte Detektion von ausgewählten strukturellen Veränderungen in den Genen
ALK, RET, ROS1, FGFR2, FGFR3, NTRK1
Weitere Informationen
Webinar: Discover the Power of Modern Tumor Diagnostics
Liquid biopsy in der genetischen Tumordiagnostik – CancerDetect®
Hinweis: Die Inhalte sind nur in englischer Sprache verfügbar.
Downloads
Kontaktieren Sie uns
Sie haben noch eine Frage oder Interesse an unserem Service?
Diagnostik-Support
Wir unterstützen Sie auf Wunsch bei der Auswahl der diagnostischen Strategie – für jede einzelne Patientin und jeden einzelnen Patienten.