Das Exom beinhaltet alle proteinkodierenden Regionen des menschlichen Genoms, die auch als Exons bezeichnet werden. Obwohl Exons nur 1 % bis 2 % des Genoms ausmachen, befinden sich circa 89 % aller bekannten krankheitsverursachenden Mutationen in diesen Regionen. Allerdings können krankheitsrelevante Varianten auch in nicht-kodierenden Regionen vorkommen. Wir haben ExomeXtra® Sequencing entwickelt, um auch die krankheitsrelevanten Varianten in nicht-kodierenden Regionen zu untersuchen, die in der Human Gene Mutation Database (HGMD) und der ClinVar-Datenbank beschrieben sind.
Zusätzlich zu diesen nicht-kodierenden Varianten haben wir ein neues und einzigartiges CNV-Backbone in unser ExomeXtra® Sequencing aufgenommen, welches die genomweite Analyse und den hochauflösenden Nachweis von Kopienzahlveränderungen („copy number variations“, CNVs) ermöglicht. Dieses neue CNV-Backbone ermöglicht die Bestimmung von CNVs in kodierenden Regionen und die Entdeckung von CNVs in nicht-kodierenden Regionen. Dadurch erhöht es die Wahrscheinlichkeit, krankheitsverursachende Mutationen zu finden.
Unsere ExomeXtra®-Sequencing-Produkte kombinieren die Vorteile der Sequenzierung des gesamten Exoms („whole exome sequencing“, WES) und Genoms („whole genome sequencing“, WGS). Dabei werden alle krankheitsrelevanten genomischen Regionen abgedeckt.
CeGaT ist der beste Partner für Ihr Sequenzierprojekt
Unser Engagement für Sie
Schnelle Bearbeitung
Bearbeitungszeit
≤ 15 Werktage
Hohe Qualität
Höchste Genauigkeit bei allen Prozessen
Sichere Datenlieferung
Sichere Bereitstellung der sequenzierten Daten über hausinterne Server
Sichere Aufbewahrung
Sichere Proben- und Datenaufbewahrung nach Projektabschluss
Unser Service
Uns ist eine umfangreiche und erstklassige Projektbegleitung wichtig − von der Auswahl des passenden Produktes bis zur Auswertung der Daten. Jedes Projekt wird von einer engagierten Wissenschaftlerin oder einem engagierten Wissenschaftler begleitet, so dass Ihnen während des gesamten Projektverlaufs eine Ansprechpartnerin oder ein Ansprechpartner zur Seite steht.
Unser Service umfasst:
- Ausführliche Projektberatung
- Produktauswahl abgestimmt auf Ihr Projekt
- Umfangreiche bioinformatische Auswertung Ihrer Daten
- Abschließender und detaillierter Projektbericht mit Informationen zur Probenqualität, Sequenzierparametern, bioinformatischen Analysen und Ergebnissen
Profitieren Sie von unserem hervorragenden Support und unseren akkreditierten Arbeitsabläufen.
Unser Produktportfolio für ExomeXtra® Sequencing
Wir bieten verschiedene ExomeXtra®-Sequencing (EXS)-Produkte für eine Vielzahl von Forschungsfragen an. Wünschen Sie zusätzlich zu den beinhalteten Leistungen bioinformatische Analysen Ihrer Daten? Jedes unserer Produkte kann durch weitere Dienstleistungen ergänzt werden. Wir beraten Sie gerne.
EXS Classic | EXS Premium | EXS Premium Deep |
Spezies Mensch | Spezies Mensch | Spezies Mensch |
DNA-Qualität Hochmolekulare DNA | DNA-Qualität Unterschiedliche/geringe Qualität | DNA-Qualität Unterschiedliche/geringe Qualität |
Anreicherungsmethode ExomeXtra® Sequencing | Anreicherungsmethode ExomeXtra® Sequencing | Anreicherungsmethode ExomeXtra® Sequencing |
Zielregion Alle proteinkodierenden Regionen + mitochondriales Genom | Zielregion Alle proteinkodierenden Regionen + mitochondriales Genom | Zielregion Alle proteinkodierenden Regionen + mitochondriales Genom |
Sequenzierplattform Illumina | Sequenzierplattform Illumina | Sequenzierplattform Illumina |
Output 18 Gb | Output 18 Gb | Output 24 Gb |
Beinhaltete Leistungen Projektbericht & Dateien im FASTQ-Format | Beinhaltete Leistungen Projektbericht & Dateien im FASTQ-Format | Beinhaltete Leistungen Projektbericht & Dateien im FASTQ-Format |
Bioinformatik
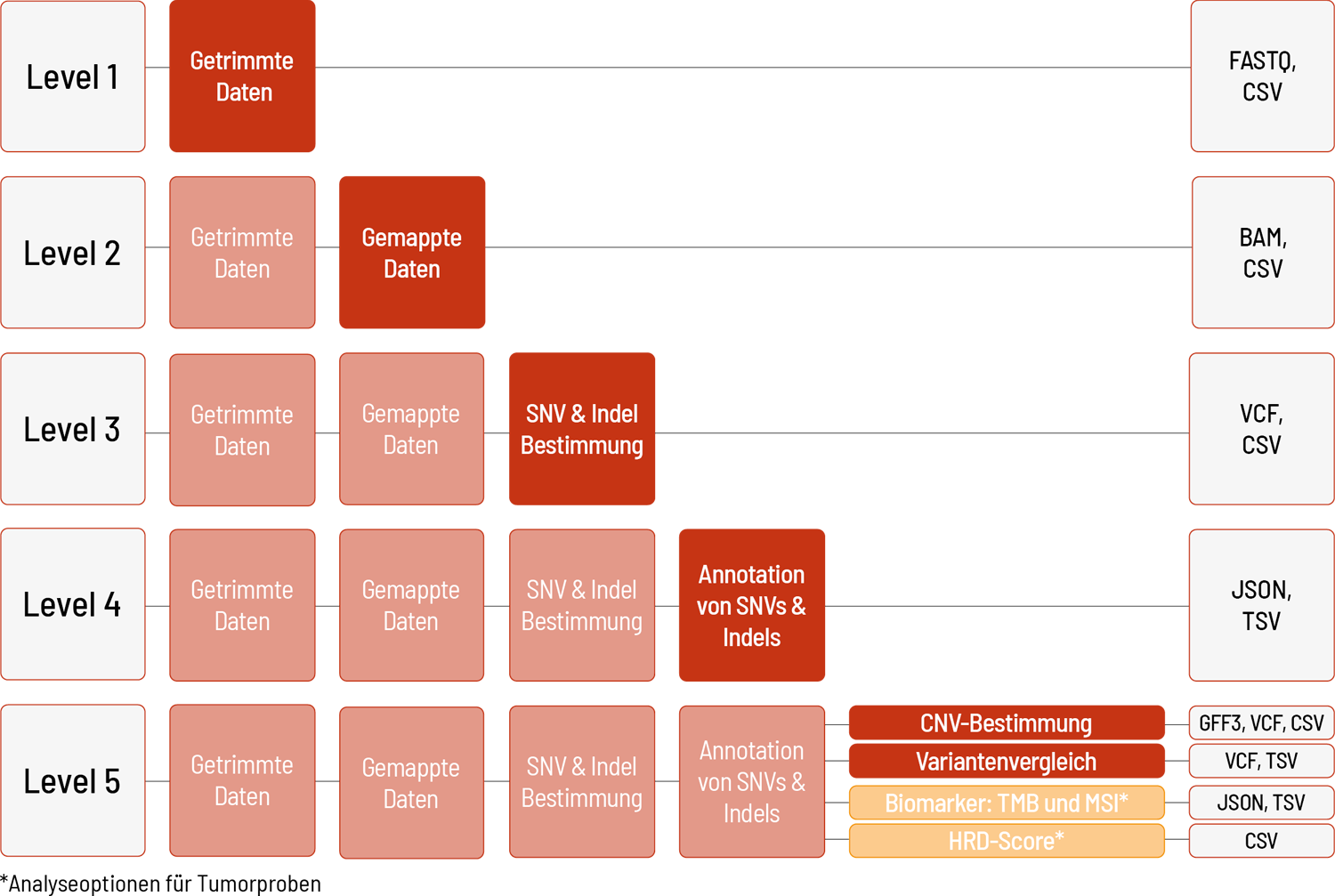
Die Rohdaten der Sequenzierung werden automatisch verarbeitet. Wir bieten verschiedene Level bioinformatischer Analysen an. Das Standardlevel ist Level 1. Mit steigendem Bioinformatiklevel werden mehr Daten geliefert. Alle höheren Level beinhalten dabei die Daten der vorherigen Level. Zusätzlich zu den Daten und unabhängig vom Analyselevel wird ein Projektbericht verfasst.
Alle EXS-Produkte werden mit der Illumina DRAGEN Bio-IT-Plattform analysiert. Wir können die Analysen basierend auf den menschlichen Referenzgenomen hg19 oder GRCh38 anbieten.
Level 1:
- Demultiplexing und Adapter-Trimming der Sequenzierdaten (FASTQ-Datei)
- Metriken (CSV-Datei)
- MultiQC-Bericht (HTML-Datei)
Level 2:
- Mapping der Sequenzierdaten (BAM-Datei)
- Metriken (CSV-Datei)
Level 3:
- Bestimmung von Einzelnukleotid-Varianten („single nucleotide variants“, SNVs) und kleinen Insertionen und Deletionen (Indels)
- Metriken (CSV-Datei)
Level 4:
- Annotationen von SNVs und Indels (JSON- und TSV-Datei) (nur für menschliche Proben)
Level 5 (eine der folgenden Möglichkeiten):
- Bestimmung von Kopienzahlveränderungen („copy number variations“, CNVs) (VCF- und GFF3-Datei) inklusive Metriken (CSV-Datei) (nur für menschliche Proben)
- Variantenvergleich mehrerer Proben, z. B. Triofilterung (VCF- und TSV-Datei)
Für Tumorproben (Tumor Only und Tumor-Normal-Vergleich) können wir zusätzlich folgende Analyse anbieten:
- Biomarker-Bestimmung: TMB und MSI (CSV- und JSON-Datei)
Für Tumorproben mit entsprechendem Normalgewebe (Tumor-Normal-Vergleich) können wir zusätzlich folgende Analyse anbieten:
- Berechnung des HRD-Scores (CSV-Datei)
Technische Information
Bei CeGaT wird die Paired-End-Sequenzierung (2 x 100 bp) mit den Sequenzierplattformen von Illumina durchgeführt.
Wir weisen darauf hin, dass unsere ExomeXtra®-Sequencing-Produkte regelmäßig aktualisiert werden, um eine konstante Verbesserung zu gewährleisten. Während dieser Updates werden, basierend auf den neusten wissenschaftlichen Erkenntnissen, diagnostisch relevante Bereiche hinzugefügt oder entfernt. Bitte beachten Sie, dass wir immer nur die aktuellste Version anbieten können. Frühere Versionen können also leider nicht angefordert werden.
Weitere Informationen zu ExomeXtra® Sequencing
Obwohl das Exom nur 1 %–2 % des Genoms ausmacht, liegen innerhalb des Exoms 89 % aller krankheitsverursachenden Mutationen. Um allerdings komplexe Krankheitsbilder zu entschlüsseln, können zusätzliche genetische Informationen nötig sein. Handelsübliche Exome decken oft krankheitsrelevante Varianten in intronischen oder regulatorischen Regionen nicht ab. Daher haben wir ExomeXtra® entwickelt, um alle bekannten pathogenen Regionen des menschlichen Genoms zu inkludieren – unabhängig von ihrer intronischen oder exonischen Lage.
Im ersten Entwicklungsschritt haben wir alle verfügbaren Technologien untersucht, um die Technologie zu finden, die uns die umfangreichste und gleichmäßigste Abdeckung aller krankheitsverursachenden Varianten liefert. Die Varianten können dabei sowohl in kodierenden als auch nicht-kodierenden Regionen des Genoms liegen. Basierend auf diesen Untersuchungen haben wir die ExomeXtra®-Anreicherung mit der besten Abdeckung aller krankheitsverursachenden Varianten entwickelt. Nach der Sequenzierung unserer ExomeXtra®-Produkte werden die qualitativ hochwertigen Daten mit unserer Bioinformatik-Pipeline in zahlreichen Analyseschritten prozessiert.
Unsere ExomeXtra®-Anreicherung wird regelmäßig basierend auf den neusten wissenschaftlichen Erkenntnissen aktualisiert. Während dieser Updates werden relevante Datenbanken, wie die HGMD-oder ClinVar-Datenbank, durchsucht. Diese Suche umfasst neue Varianten in nicht-kodierenden Bereichen, die als pathogen oder wahrscheinlich-pathogen eingestuft werden. Die Varianten werden dann in die ExomeXtra®-Anreicherung inkludiert.
Gleichzeitig werden Varianten, die nicht mehr als pathogen oder wahrscheinlich-pathogen eingestuft werden, aus der Anreicherung entfernt. Somit beinhaltet unser ExomeXtra® stets alle genomischen Regionen, die in relevanten Datenbanken als krankheitsrelevant eingestuft werden.
Mit unseren ExomeXtra®-Sequencing-Produkten können sogar Deletionen und Duplikationen im gesamten Genom nachgewiesen werden. Das neue und einzigartige CNV-Backbone ermöglicht die genomweite Analyse und den Nachweis von Kopienzahlveränderungen („copy number variations“, CNVs) mit einer hohen Auflösung. Der CNV-Nachweis sowohl in kodierenden als auch nicht-kodierenden Bereichen ist einzigartig in der Exom-Sequenzierung. Unser ExomeXtra® Sequencing bietet eine gleichmäßige Abdeckung und eine höhere Wahrscheinlichkeit, krankheitsverursachende Mutationen zu entdecken.
Unser internes Design des ExomeXtra® berücksichtigt klinisch relevante Abschnitte von nicht-kodierenden RNA-Genen, flankierende intronische Sequenzen und das gesamte mitochondriale Genom. Daher ist das ExomeXtra® Sequencing eine gute Möglichkeit, die genetische Ursache einer Erkrankung zu finden.
Downloads
Kontaktieren Sie uns
Sie haben noch Fragen oder Interesse an unserem Service? Treten Sie gern mit uns in Kontakt. Wir werden uns schnellstmöglich um Ihr Anliegen kümmern.
Starten Sie Ihr Projekt mit uns
Gerne beraten wir Sie zu unseren Sequenzierdienstleitungen und erarbeiten mit Ihnen gemeinsam die beste Lösung, die auf Ihre klinische Studie oder Forschungsprojekt abgestimmt ist.
Bitte geben Sie, falls möglich, folgende Probeninformationen an: Ausgangsmaterial, Anzahl der Proben, bevorzugte Option für die Vorbereitung der Library, bevorzugte Sequenziertiefe und gewünschte bioinformatische Analysestufe.