Die Präzisionsmedizin bringt einen bedeutenden Paradigmenwechsel in die Arzneimittelforschung und Therapie von Tumorerkrankungen. Bisher nutzte die Medizin einheitliche Behandlungsstrategien für alle. Ein umfassendes genomisches Tumorprofiling (“comprehensive genomic tumor profiling”, CGP) ist inzwischen von außerordentlicher Bedeutung. Ein umfassendes Tumorprofiling bezieht sich auf die gleichzeitige Auswertung mehrerer Biomarker in einer Analyse. Mit diesem Ansatz lassen sich nicht nur die wichtigsten prädiktiven Marker für aktuelle, zielgerichtete Therapien, sondern auch wichtige immunonkologische Biomarker wie die Tumormutationslast (“tumor mutational burden”, TMB) und die Mikrosatelliteninstabilität (“microsatellite instability”, MSI) ermitteln. Der TMB ist ein Biomarker, der die Anzahl der somatischen Mutationen im Tumor einer Krebspatientin oder eines Krebspatienten misst und als Mutationen pro Megabase (mut/Mb) quantifiziert wird. Ein weiterer wichtiger Biomarker für die Immuntherapie ist die MSI, die durch das Versagen des DNA-Fehlpaarungsreparatur-Systems (“DNA mismatch repair system”) verursacht wird.
Die Anwendungsbereiche des umfassenden Tumorprofilings sind vielfältig, mit besonderem Fokus auf:
- Stratifizierung von Patientinnen und Patienten für die beste Behandlungswahl
- Identifizierung von Patientinnen und Patienten, die für klinische Studien in Frage kommen
- Förderung der klinischen Forschung, insbesondere auf dem Gebiet der Immuntherapie
Wählen Sie zwischen verschiedenen Produkten, um ein umfassendes Tumorprofiling durchzuführen.
CeGaT ist der beste Partner für Ihr Sequenzierprojekt
Unser Engagement für Sie
Schnelle Bearbeitung
Bearbeitungszeit
≤ 15 Werktage
Hohe Qualität
Höchste Genauigkeit bei allen Prozessen
Sichere Datenlieferung
Sichere Bereitstellung der sequenzierten Daten über hausinterne Server
Sichere Aufbewahrung
Sichere Proben- und Datenaufbewahrung nach Projektabschluss
Unser Service
Uns ist eine umfangreiche und erstklassige Projektbegleitung wichtig − von der Auswahl des passenden Produktes bis zur Auswertung der Daten. Jedes Projekt wird von einer engagierten Wissenschaftlerin oder einem engagierten Wissenschaftler begleitet, so dass Ihnen während des gesamten Projektverlaufs eine Ansprechpartnerin oder ein Ansprechpartner zur Seite steht.
Unser Service umfasst:
- Ausführliche Projektberatung
- Produktauswahl abgestimmt auf Ihr Projekt
- Umfangreiche bioinformatische Auswertung Ihrer Daten
- Abschließender und detaillierter Projektbericht mit Informationen zur Probenqualität, Sequenzierparametern, bioinformatischen Analysen und Ergebnissen
Profitieren Sie von unserem hervorragenden Support und unseren akkreditierten Arbeitsabläufen.
Unser Produktportfolio für Comprehensive Tumor Profiling
Wir bieten verschiedene Comprehensive-Tumor-Profiling (CTP)-Produkte für eine Vielzahl von Forschungsfragen an. Unsere CTP-Somatic- und CTP-Tumor-Produkte basieren auf der Anreicherung unserer Whole-Exome-Sequencing (WES)-Produkte und werden analog zu WES mit der DRAGEN Bio-IT-Plattform analysiert. Diese Analysen bieten Informationen über mehr als 20.000 Gene. Für CTP Somatic werden Proben von Tumor- und Normalgewebe für den Tumor-Normal-Vergleich benötigt. Für CTP Tumor benötigen wir lediglich eine Tumorprobe.
Sollten Sie nur an einem Teil der Gene interessiert sein, die für das Profiling von Tumoren relevant sind, bieten wir verschiedene Panel-Sequencing-Produkte innerhalb unseres Comprehensive-Tumor-Profiling-Produktportfolios an. Das CTP TUM Panel deckt mehr als 760 krebsassoziierte Gene ab. Darüber hinaus werden 31 therapierelevante Fusionen mit einer hohen Sequenziertiefe analysiert. Unser CTP PAT Panel deckt 55 Gene ab, die relevant für Tumorerkrankungen sind. Zusätzlich bieten wir das CTP FUS Panel an. Im Gegensatz zu den anderen beiden DNA-Panels handelt es sich beim CTP FUS Panel um ein RNA-Panel, das sich auf Fusionen konzentriert, die relevant für Tumorerkrankungen sind.
Wünschen Sie zusätzlich zu den beinhalteten Leistungen bioinformatische Analysen Ihrer Daten? Jedes unserer Produkte kann durch weitere Dienstleistungen ergänzt werden. Wir beraten Sie gerne.
CTP Somatic | CTP Tumor | CTP TUM Panel | CTP PAT Panel | CTP FUS Panel |
Spezies | Spezies | Spezies | Spezies | Spezies |
Sequenzierpanel | Sequenzierpanel | Sequenzierpanel | Sequenzierpanel | Sequenzierpanel |
Anzahl analysierter Gene | Anzahl analysierter Gene | Anzahl analysierter Gene | Anzahl analysierter Gene | Anzahl analysierter Gene |
Analyse von Tumor- und Normalgewebe | Analyse von Tumor- und Normalgewebe | Analyse von Tumor- und Normalgewebe | Analyse von Tumor- und Normalgewebe | Analyse von Tumor- und Normalgewebe |
Ausgangsmaterial | Ausgangsmaterial | Ausgangsmaterial | Ausgangsmaterial | Ausgangsmaterial |
Sequenzierplattform | Sequenzierplattform | Sequenzierplattform | Sequenzierplattform | Sequenzierplattform |
Output | Output (abhängig vom Tumorgehalt) | Output | Output | Output |
Beinhaltete Leistungen | Beinhaltete Leistungen | Beinhaltete Leistungen | Beinhaltete Leistungen | Beinhaltete Leistungen |
Wenn Sie sich für Sequenzierpanels unabhängig vom Tumorprofiling interessieren, könnte unser Panel-Sequencing-Produktportfolio interessant für Sie sein.
Bioinformatik
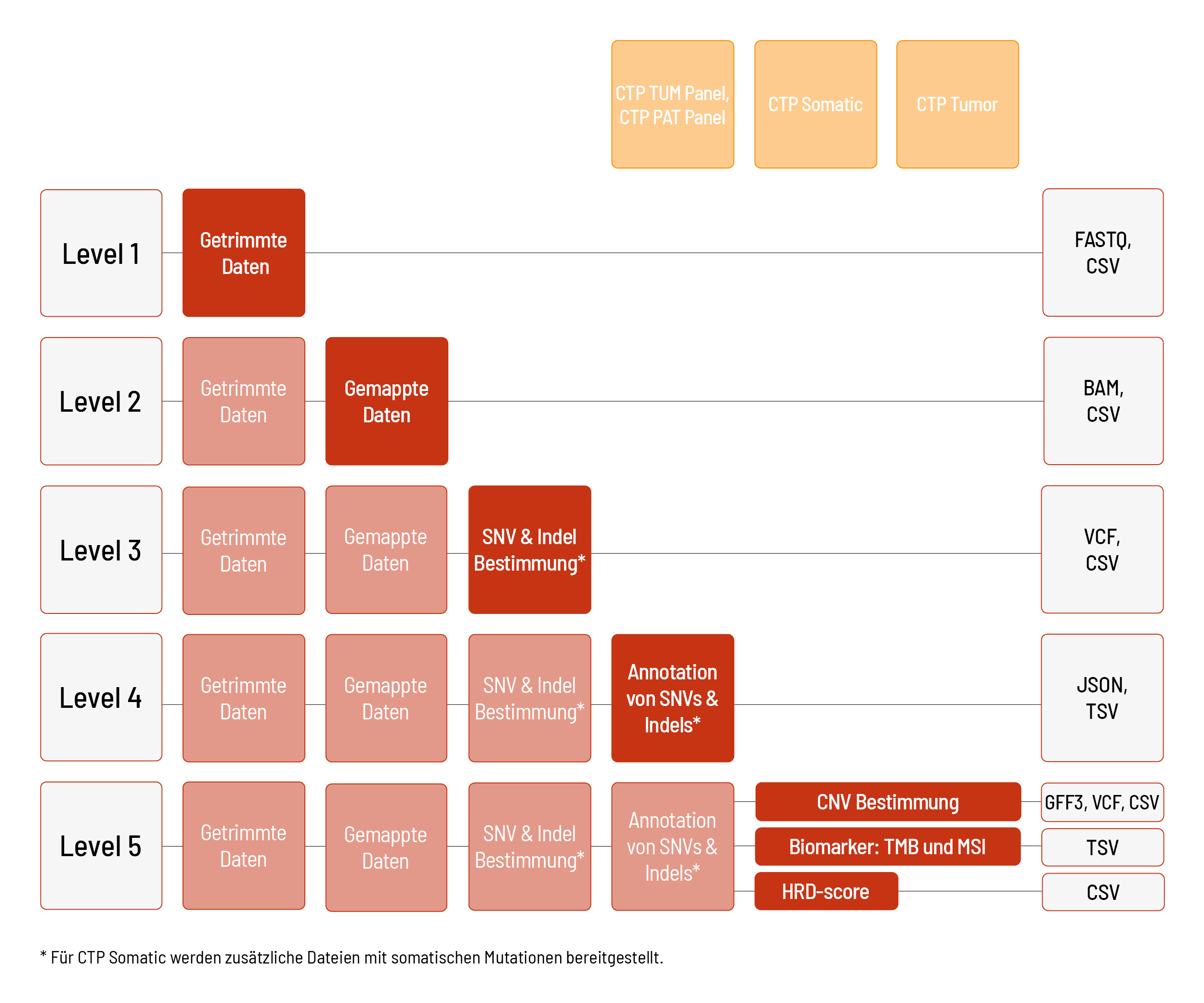
Die Rohdaten der Sequenzierung werden automatisch verarbeitet. Wir bieten verschiedene Level bioinformatischer Analysen an. Das Standardlevel ist Level 1. Mit steigendem Bioinformatiklevel werden mehr Daten geliefert. Alle höheren Level beinhalten dabei die Daten der vorherigen Level. Zusätzlich zu den Daten und unabhängig vom Analyselevel wird ein Projektbericht verfasst.
Analysen für CTP Somatic, CTP Tumor, CTP TUM Panel und CTP PAT Panel:
Wir können die Analysen für CTP Somatic und CTP Tumor basierend auf den menschlichen Referenzgenomen hg19 oder GRCh38 anbieten. Die Analysen für CTP TUM Panel und CTP PAT Panel basieren auf dem menschlichen Referenzgenom hg19 und sind bis Level 4 verfügbar.
Level 1:
- Demultiplexing und Adapter-Trimming der Sequenzierdaten (FASTQ-Datei)
- Metriken (CSV-Datei)
- MultiQC-Bericht (HTML-Datei)
Level 2:
- Mapping der Sequenzierdaten (BAM-Datei)
- Metriken (CSV-Datei)
Level 3:
- Bestimmung von Einzelnukleotid-Varianten (“single nucleotide variants”, SNVs) und kleinen Insertionen und Deletionen (kurz Indels) (VCF-Datei)
- Metriken (CSV-Datei)
Level 4:
- Annotation von SNVs und Indels (JSON- und TSV-Datei)
Level 5 (eine der folgenden Möglichkeiten):
- Bestimmung von Kopienzahlveränderungen (“copy number variations”, CNVs) (VCF- und GFF3-Datei) inklusive Metriken (CSV-Datei)
- Biomarker-Bestimmung: TMB und MSI (TSV-Datei)
- Berechnung des HRD-Scores (CSV-Datei) (nur für CTP Somatic)
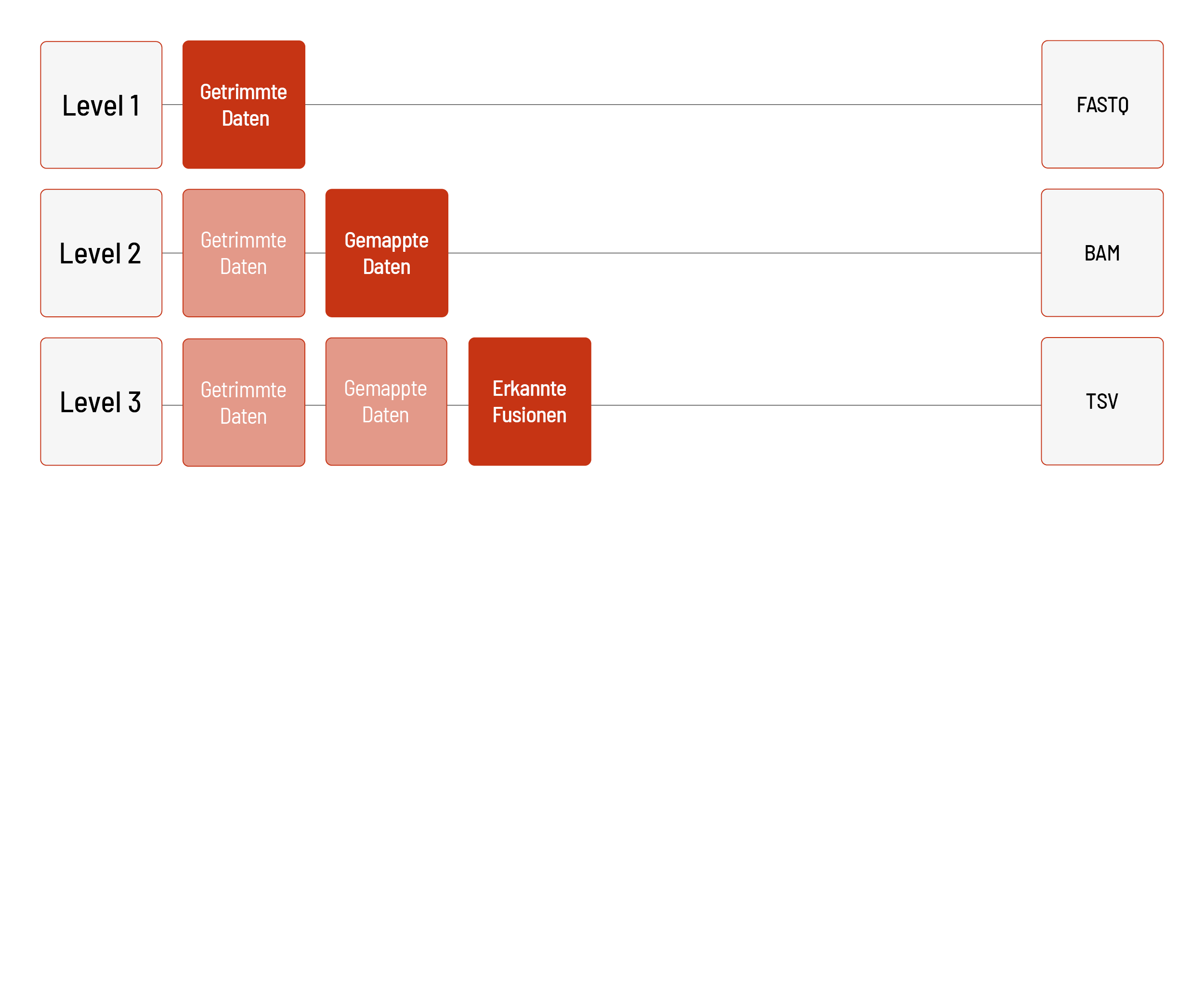
Analysen für CTP FUS Panel:
Wir können die Analysen für CTP FUS Panel basierend auf dem menschlichen Referenzgenom hg19 anbieten.
Level 1:
- Demultiplexing und Adapter-Trimming der Sequenzierdaten
(FASTQ-Datei)
Level 2:
- Mapping der Sequenzierdaten (BAM-Datei)
Level 3:
- Lieferung der erkannten Fusionen (TSV-Datei)
Technische Information
Bei CeGaT wird die Paired-End-Sequenzierung (2 x 100 bp) mit den Sequenzierplattformen von Illumina durchgeführt. Wenn Sie andere Sequenzierparameter benötigen, lassen Sie es uns gerne wissen! Wir können Ihnen weitere Lösungen anbieten.
Genverzeichnis für CTP TUM Panel, CTP PAT Panel und CTP FUS Panel
Weitere Informationen zu Comprehensive Tumor Profiling
Die Tumormutationslast (“tumor mutational burden”, TMB) beschreibt die Anzahl der Genmutationen, die im Tumor, aber nicht im gesunden Gewebe einer erkrankten Person auftreten. Bei diesen Mutationen handelt es sich also um kodierende, somatische Mutationen. Die Tumormutationslast ist definiert als Mutationen pro Megabase (mut/Mb). Sie ist nicht zu verwechseln mit der Varianten-Allel-Häufigkeit, die lediglich die Häufigkeit eines Allels angibt. Im Gegensatz dazu ist die Tumormutationslast die Gesamtmenge der Mutationen pro Megabasen in einer Probe. Je nach Tumorentität variiert diese Mutationslast: Einige Tumoren weisen keine hohen Mutationsraten auf, wie z. B. das Ewing-Sarkom oder die akute myeloische Leukämie. Andere Krebsarten, wie Melanome oder Plattenepithelkarzinome der Lunge, weisen eine hohe Mutationslast des Tumors auf. Anhand der Tumormutationslast kann der Tumor also als niedrig, mittel oder hoch mutiert eingestuft werden. Die Tumormutationslast ist ein vielversprechender prädiktiver und immuntherapeutischer Biomarker, zum Beispiel für die Immun-Checkpoint-Inhibitor-Therapie. Eine solche Immuntherapie ist bei Patientinnen und Patienten mit einer hohen Tumormutationslast besonders wirksam. Eine höhere Anzahl tumorspezifischer Mutationen führt zu einer höheren Anzahl veränderter Proteine, so genannter Neoantigene. Diese Neoantigene unterscheiden sich von den gesunden Proteinen, was zu einer stärkeren Differenzierung des Tumorgewebes gegenüber dem gesunden Gewebe führt.
Sowohl Tumorzellen als auch gesunde Zellen präsentieren Teile ihres Proteingehalts auf ihrer Zelloberfläche. Diese kurzen, präsentierten Proteinteile werden auch Peptide genannt. Immunzellen überprüfen regelmäßig die Peptide auf den Oberflächen der Proteine. Wenn die Peptide als fremd erkannt werden, wird das Immunsystem aktiviert. Die Tumorpeptide, also die Neoantigene, sind für das Immunsystem fremd. Daher korreliert eine höhere Mutationslast des Tumors mit mehr Mutationen und somit auch mit mehr Neoantigenen, die vom Immunsystem als fremd erkannt werden. Bei einer höheren Mutationslast des Tumors wird also das Immunsystem leichter aktiviert.
Ein weiterer immuntherapeutischer Biomarker ist die Mikrosatelliteninstabilität (“microsatellite instability”, MSI). Die Mikrosatelliteninstabilität zeigt die Anzahl der Mutationen in so genannten Satelliten-Regionen an. Bei diesen Regionen handelt es sich um spezielle Wiederholungssequenzen in der DNA. Die Satelliten-Regionen der DNA sind der Hauptbestandteil der funktionellen Zentromere und bilden die Hauptstruktur des Heterochromatins. Ein gestörter DNA-Mismatch-Repair-Mechanismus, kurz MMR, kann zu einer genetischen Hypermutationsfähigkeit in den Satelliten-Regionen führen, was eine Mikrosatelliteninstabilität zur Folge hat. Die Mikrosatelliteninstabilität wird bei einem niedrigen MSI-Score als stabil und bei einem hohen MSI-Score als instabil eingestuft.
Downloads
Kontaktieren Sie uns
Sie haben noch Fragen oder Interesse an unserem Service? Treten Sie gern mit uns in Kontakt. Wir werden uns schnellstmöglich um Ihr Anliegen kümmern.
Starten Sie Ihr Projekt mit uns
Gerne beraten wir Sie zu unseren Sequenzierdienstleitungen und erarbeiten mit Ihnen gemeinsam die beste Lösung, die auf Ihre klinische Studie oder Forschungsprojekt abgestimmt ist.
Bitte geben Sie, falls möglich, folgende Probeninformationen an: Ausgangsmaterial, Anzahl der Proben, bevorzugte Option für die Vorbereitung der Library, bevorzugte Sequenziertiefe und gewünschte bioinformatische Analysestufe.