Wissenschaftliche Studien belegen einen engen Zusammenhang zwischen der Zusammensetzung des menschlichen Mikrobioms und dem Auftreten verschiedener Krankheiten oder der Reaktion auf Arzneimittel. Vor allem für das Darmmikrobiom wurde dieser Zusammenhang nachgewiesen. Aber auch die Mikrobiome anderer Körperregionen, wie der Haut, dem Mund oder der Nase können eine Rolle bei der Entstehung von Erkrankungen spielen. Darüber hinaus sind Mikroorganismen an vielen biochemischen Reaktionen in der Umwelt beteiligt und stellen wichtige Bestandteile von Ökosystemen dar. Das Verständnis der mikrobiellen Funktionen in spezifischen Mikrobiomen oder in Wirt-Mikroben-Beziehungen bietet ein großes Potenzial für neue, therapeutische Entdeckungen, insbesondere für die Mikroorganismen, die schwer zu kultivieren sind.
Mithilfe von Shotgun Metagenomic Sequencing wird der gesamte DNA-Gehalt einer Probe analysiert und ermöglicht den Nachweis von Mikroben (Bakterien, Archaeen, Pilzen, Protozoen, Viren usw.) bis auf die Artenebene genau. Dementsprechend können funktionelle Gene untersucht werden, die für bestimmte Stoffwechselenzyme kodieren. Daher ist für eine gründliche Charakterisierung von Mikrobiomen, einschließlich der genauen Identifizierung der Arten und ihrer Funktionen, Shotgun Metagenomic Sequencing die beste Wahl.
Die Anwendungen von Shotgun Metagenomic Sequencing sind vielfältig und decken unter anderem folgende Bereiche ab:
- Krankheitsüberwachung
- Nachweis von mikrobiellen Biomarkern
- Entwicklung von Arzneimitteln
- Charakterisierung von Umweltmikrobiomen
- Entdeckung neuer mikrobieller Arten (de novo-Assemblierung)
Wir bieten verschiedene Shotgun-Metagenomic-Sequencing-Produkte an, um eine Vielzahl von Forschungsfragen zu beantworten.
CeGaT ist der beste Partner für Ihr Sequenzierprojekt
Unser Engagement für Sie
Schnelle Bearbeitung
Bearbeitungszeit
≤ 15 Werktage
Hohe Qualität
Höchste Genauigkeit bei allen Prozessen
Sichere Datenlieferung
Sichere Bereitstellung der sequenzierten Daten über hausinterne Server
Sichere Aufbewahrung
Sichere Proben- und Datenaufbewahrung nach Projektabschluss
Unser Service
Uns ist eine umfangreiche und erstklassige Projektbegleitung wichtig − von der Auswahl des passenden Produktes bis zur Auswertung der Daten. Jedes Projekt wird von einer engagierten Wissenschaftlerin oder einem engagierten Wissenschaftler begleitet, so dass Ihnen während des gesamten Projektverlaufs eine Ansprechpartnerin oder ein Ansprechpartner zur Seite steht.
Unser Service umfasst:
- Ausführliche Projektberatung
- Produktauswahl abgestimmt auf Ihr Projekt
- Umfangreiche bioinformatische Auswertung Ihrer Daten
- Abschließender und detaillierter Projektbericht mit Informationen zur Probenqualität, Sequenzierparametern, bioinformatischen Analysen und Ergebnissen
Profitieren Sie von unserem hervorragenden Support und unseren akkreditierten Arbeitsabläufen.
Unser Produktportfolio für Shotgun Metagenomic Sequencing
Wir bieten verschiedene Shotgun-Metagenomic-Sequencing-Produkte für eine Vielzahl von Forschungsfragen an. Wünschen Sie zusätzlich zu den beinhalteten Leistungen bioinformatische Analysen Ihrer Daten? Jedes unserer Produkte kann durch weitere Dienstleistungen ergänzt werden. Wir beraten Sie gerne.
Shotgun Metagenomics Classic | Shotgun Metagenomics Flex |
Spezies | Spezies |
Probenart | Probenart |
Ziel | Ziel |
PCR-Amplifikation | PCR-Amplifikation |
Read-Länge | Read-Länge |
Sequenzierplattform | Sequenzierplattform |
Output | Output |
Beinhaltete Leistungen | Beinhaltete Leistungen |
Bioinformatik
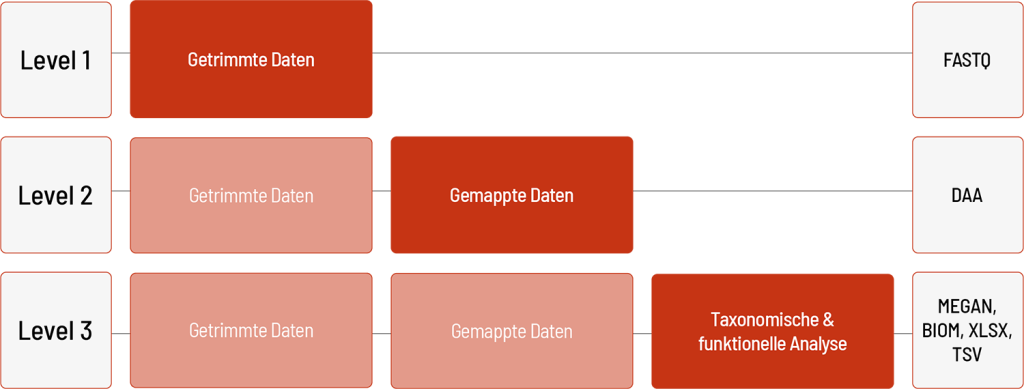
Die Rohdaten der Sequenzierung werden automatisch verarbeitet. Wir bieten verschiedene Level bioinformatischer Analysen an. Das Standardlevel ist Level 1. Mit steigendem Bioinformatiklevel werden mehr Daten geliefert. Alle höheren Level beinhalten dabei die Daten der vorherigen Level. Zusätzlich zu den Daten und unabhängig vom Analyselevel wird ein Projektbericht verfasst.
Level 1:
- Demultiplexing und Adapter-Trimming der Sequenzierdaten (FASTQ-Datei)
Level 2:
- Mapping der Sequenzierdaten gegen die Mikriobiom-Datenbank (DAA-Datei*)
Level 3:
- Taxonomische und funktionelle Klassifizierung mit MEGAN 6 (MEGAN-Datei*)
- Erstellung von taxonomischen Häufigkeitstabellen mit Anzahl der Sequenzen und relativen Häufigkeiten bis hinunter zur Artenebene (XLSX- und BIOM-Datei)
- Erstellung von Tabellen mit mikrobiellen Funktionen, einschließlich der Anzahl der Sequenzen und der relativen Häufigkeiten der funktionellen Gene, die mit Hilfe der KEGG-Datenbank klassifiziert wurden (XLSX-Datei)
- Erstellung von Balkendiagrammen, die die relativen Häufigkeiten bis auf die Artenebene zeigen (PNG-Datei)
* Diese Dateien können mit MEGAN6, einer benutzerfreundlichen und kostenlosen Software zur Analyse von Mikrobiomdaten, interaktiv untersucht und weiter analysiert werden.
Technische Information
Bei CeGaT wird die Paired-End-Sequenzierung (2 x 100 bp) mit den Sequenzierplattformen von Illumina durchgeführt. Wenn Sie andere Sequenzierparameter benötigen, lassen Sie es uns gerne wissen! Wir können Ihnen weitere Lösungen anbieten.
Wir freuen uns, Ihnen unser speziell entwickeltes Stuhlproben-Kit anbieten zu können. Dieses ermöglicht eine bequeme Entnahme von fäkalem Material. In Kombination mit unserem DNA-Isolationsservice kommt es dadurch nicht zu verfälschten Ergebnissen. Bitte setzen Sie sich mit uns in Verbindung, um die Probenahme und DNA-Extraktion aus verschiedenen Ausgangsmaterialien zu besprechen.
Weitere Informationen zu Shotgun Metagenomic Sequencing
Bei der so genannten Shotgun-Metagenom-Sequenzierung, auch bekannt als Shotgun Metagenomic Sequencing, wird das gesamte Genom aller Organismen in einer Probe sequenziert. Im Gegensatz dazu wird bei der 16S-Sequenzierung nur ein Gen sequenziert. Daher ist Shotgun Metagenomic Sequencing mit der vollständigen Genomsequenzierung („whole genome sequencing“) vergleichbar. Allerdings werden bei Shotgun Metagenomic Sequencing nicht ein, sondern viele Organismen gleichzeitig sequenziert. Das Besondere an Shotgun Metagenomic Sequencing ist die Vielseitigkeit. Neben Bakterien und Archaeen können auch Eukaryoten und Viren identifiziert werden. Shotgun Metagenomic Sequencing kann zur Analyse des Darmmikrobioms verwendet werden. Aber auch andere Körperregionen, wie die Mund- oder Nasenhöhle, besitzen ein Mikrobiom, das analysiert werden kann. Doch nicht nur menschliche Körperregionen können mit Shotgun Metagenomic Sequencing untersucht werden: Auch Umweltproben, wie z. B. Bodenproben können analysiert werden. Mit Shotgun Metagenomic Sequencing können alle Organismen einer Probe taxonomisch bis auf Artenebene differenziert werden. Darüber hinaus ist eine funktionelle Analyse möglich. Bei einer ausreichenden Anzahl von Reads ist die Shotgun-Metagenom-Sequenzierung im Vergleich zur 16S-Sequenzierung leistungsfähiger bei der Identifizierung seltener Taxa.
Für Shotgun Metagenomic Sequencing wird die DNA aus allen Zellen der Probe extrahiert. Diese wird dann in kleine Stücke fragmentiert, die unabhängig voneinander sequenziert werden. Im Gegensatz zur 16S-Sequenzierung ist kein spezifischer genetischer Locus als Zielgen erforderlich. Die sequenzierten Fragmente können an unterschiedliche Genome in der Probe aligniert werden. Diese unterschiedlichen Genome sind nicht auf bakterielle Genome beschränkt. Für das Alignment der sequenzierten Fragmente werden die 16S rRNA-Sequenz und biologisch informative Sequenzen verwendet.
Die Shotgun-Metagenomanalyse kann auch zur Messung der taxonomischen Vielfalt einer Probe verwendet werden.
Zur Bestimmung dieser taxonomischen Vielfalt gibt es verschiedene Ansätze:
- Bei der so genannten Markergen-Analyse wird eine Sequenz mit einer Datenbank bekannter taxonomisch informativer Sequenzen verglichen. Diese taxonomisch informativen Sequenzen werden auch als Markergene bezeichnet. Wenn die Sequenz ein Homolog des Markergens ist, klassifiziert und annotiert ein Algorithmus die Ähnlichkeit der Sequenz.
- Ein anderer Ansatz ist das so genannte Binning, bei dem die Sequenzen in Gruppen zusammengefasst werden. Bei der Methode des Compositional Binnings werden die Metagenom-Sequenzen in Gruppen geclustert. Die Gruppierung basiert auf der Sequenz-Zusammensetzung. Beim Similarity Binning werden die Sequenzen auf der Grundlage ihrer Ähnlichkeit mit den taxonomisch annotierten Sequenzen geclustert. Beim Fragment Recruitment Binning werden die Sequenzen an andere, nahezu identische Sequenzen angeglichen. Diese Binning-Methode liefert eine metagenomische Abschätzung des Genoms.
Durch die Analyse der taxonomischen Vielfalt einer Probe mittels Shotgun Metagenomic Sequencing können Mikrobiome genau charakterisiert werden. Hierbei kann die Shotgun-Metagenom-Sequenzierung helfen:
- bei der Überwachung von Krankheiten, die mit einem veränderten Mikrobiom einhergehen,
- beim Nachweis mikrobieller Biomarker,
- bei der Entwicklung von Arzneimitteln,
- bei der Charakterisierung von Umweltmikrobiomen
- und bei der Entdeckung neuer Mikrobenarten.
Shotgun Metagenomic Sequencing erweitert das Wissen und Verständnis über die Funktionen und die Charakterisierung spezifischer Stämme, was zu therapeutischen Entdeckungen und innovativen Wegen zur Synthese neuer Produkte führt.
Downloads
Kontaktieren Sie uns
Sie haben noch Fragen oder Interesse an unserem Service? Treten Sie gern mit uns in Kontakt. Wir werden uns schnellstmöglich um Ihr Anliegen kümmern.
Starten Sie Ihr Projekt mit uns
Gerne beraten wir Sie zu unseren Sequenzierdienstleitungen und erarbeiten mit Ihnen gemeinsam die beste Lösung, die auf Ihre klinische Studie oder Forschungsprojekt abgestimmt ist.
Bitte geben Sie, falls möglich, folgende Probeninformationen an: Ausgangsmaterial, Anzahl der Proben, bevorzugte Option für die Vorbereitung der Library, bevorzugte Sequenziertiefe und gewünschte bioinformatische Analysestufe.