Genfusionen sind häufig Treiber von Krebserkrankungen. Daher werden bei der Behandlung von Tumorpatientinnen und Tumorpatienten zunehmend Medikamente eingesetzt, die gezielt gegen diese Genfusionen gerichtet sind.
CeGaT hat mit CancerFusionRx® eine einzigartige Methode entwickelt, die auf RNA-Ebene strukturelle Veränderungen (Genfusionen) nachweisen kann. Diese Methode ist den DNA-basierten Methoden und auch Ansätzen auf Transkriptomebene weit überlegen und ermöglicht den Nachweis sämtlicher bekannter sowie neuartiger Genfusionen. Basierend auf den gefundenen Genfusionen können wir konkrete Medikamentenempfehlungen angeben. CancerFusionRx® ist somit ein wertvolles Instrument in der genetischen Tumordiagnostik und unterstützt Sie bei der Wahl der bestmöglichen Therapie.
Sie sind in Deutschland versichert? Unsere Kolleginnen und Kollegen vom Zentrum für Humangenetik Tübingen beraten Sie gerne!
Was wir Ihnen mit diesem Service bieten
Unser Versprechen an Sie
Service Details
- Mehr als 200 Gene für den Nachweis von Fusionen und über 120 Exon-Exon spezifische Anreicherungen wiederkehrender Bruchpunkte
- Auflistung aller in Frage kommenden Medikamente mit EMA und/oder FDA-Zulassung, für deren Anwendungsoption entsprechende Genfusionen im Tumor nachgewiesen werden konnten — mehr erfahren
Die Ergebnisse werden in einem medizinischen Befund zusammengefasst. Dieser beinhaltet eine Liste aller identifizierten klinisch relevanten strukturellen Veränderungen. Die Fusionen werden mit Gennamen, funktioneller Klasse, Transkript-ID und Einfluss auf die Proteinfunktion annotiert. Jeder einzelne medizinische Befund wird von unserem interdisziplinären Expertenteam verfasst und diskutiert. Damit garantieren wir die höchste Qualität.
Methode
Aus dem Tumormaterial isolierte RNA wird in cDNA umgeschrieben und mit einem optimierten Anreicherungsprotokoll für die Sequenzierung auf dem NovaSeq™ 6000 vorbereitet. Das Bait-Design ermöglicht dabei sowohl den gezielten Nachweis bekannter relevanter Fusionsgene sowie die Detektion von noch nicht beschriebenen Fusionen, sofern ein Fusionspartner Teil der Anreicherung ist. Zudem können innergenische Transkriptvarianten wie z. B. das MET Exon 14 skipping und aktivierende EGFR-Deletionen mit therapeutischer Relevanz nachgewiesen werden.
Anschließend wertet unser Team aus Wissenschaftlerinnen und Wissenschaftlern sowie Fachärztinnen und Fachärzten für Humangenetik die Daten aus und erstellt einen medizinischen Befund.
Beispielbefund
Allgemeine Informationen
Material
Anforderungen an das Probenmaterial für alle Analysen (mindestens 20 % Tumoranteil):
- FFPE-Tumorblock (empfohlene Probenart) oder
- FFPE-Tumorgewebe-Objektträger (mindestens 10 Slides)
- Sofern möglich: H&E-gefärbte Objektträger mit deutlich markiertem Tumorbereich. Bitte geben Sie den Tumorgehalt des markierten Tumorbereichs an.
Hier finden Sie weitere Informationen zum sicheren Versand Ihrer Probe.
Dauer
- Dauer der Untersuchung: 2–3 Wochen
Für weitere Informationen kontaktieren Sie uns bitte über das Kontaktformular oder schreiben Sie uns eine E-Mail an tumor@cegat.de.
Das macht unseren CancerFusionRx®-Service besonders
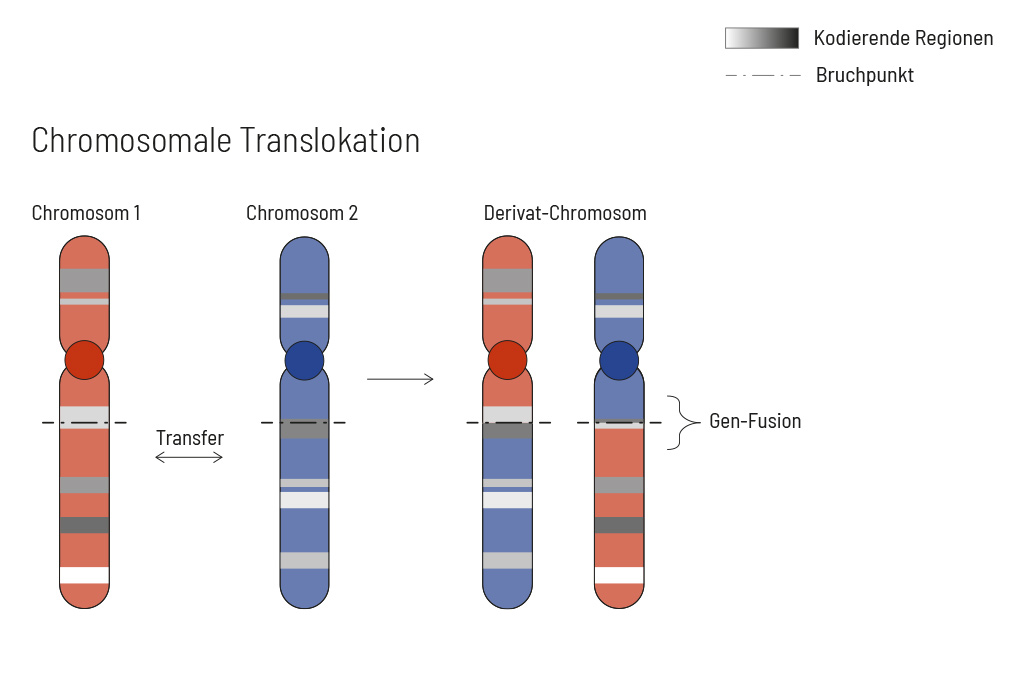
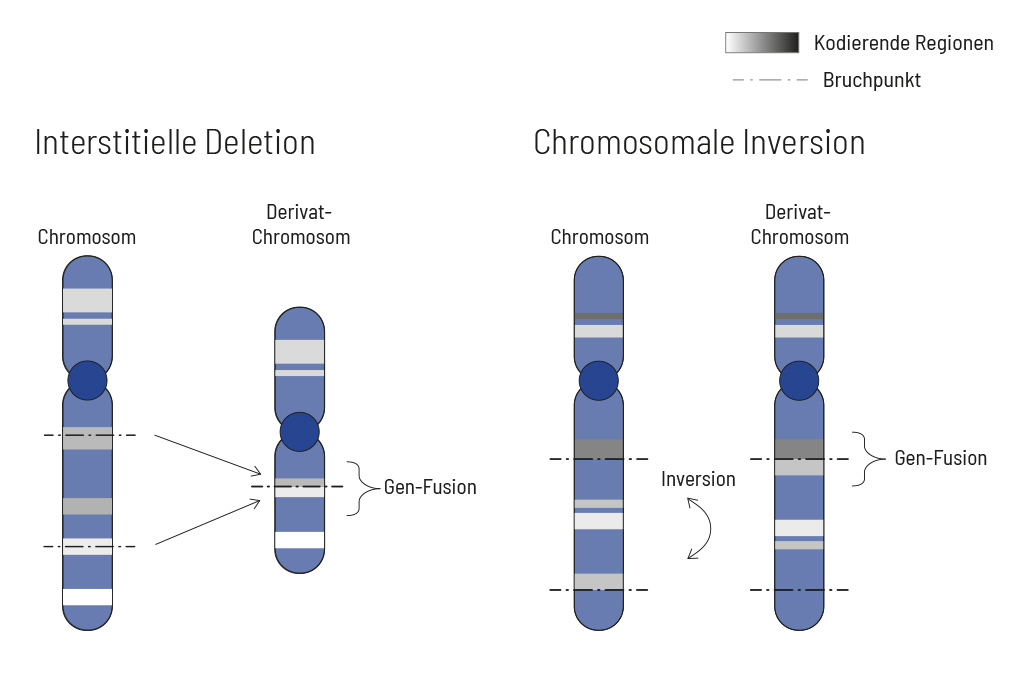
Chromosomale Veränderungen treten häufig und unabhängig von der Tumorentität auf. Infolgedessen kann es zu Genfusionen im Tumorgenom kommen. Fusionen sind oftmals starke Treiber der Tumorerkrankung und daher für Behandlungsentscheidungen sehr wichtig. Herkömmliche, auf PCR-Technologie basierende Methoden, können eine Fusion nicht nachweisen, wenn der Fusionspartner nicht bekannt ist (dies ist bei NTRK-Fusionen häufig der Fall). Selbst Analysen des gesamten Transkriptoms sind nicht sensitiv genug, insbesondere wenn der Tumorgehalt niedrig ist. Um alle bekannten und zuvor beschriebenen, sowie neuartigen Genfusionen mit einer therapeutischen Option nachzuweisen, haben wir eine gezielte Anreicherung auf RNA-Basis entwickelt.
Das Design umfasst aktuell mehr als 200 Gene für den Nachweis von Fusionen und beinhaltet über 120 Exon-Exon spezifische Anreicherungen mit bekannten Bruchpunkten. Diese Methode ist den DNA-basierten Methoden und auch Ansätzen auf Transkriptom-Basis überlegen. Wir empfehlen dringend, die genetische Tumordiagnostik durch RNA-Anreicherung für Fusionen zu ergänzen, um ein möglichst vollständiges Verständnis der Tumorbiologie zu erhalten.
Strukturelle Varianten mit potenzieller therapeutischer Relevanz
Wegweiser für potenziell wirksame Medikamente
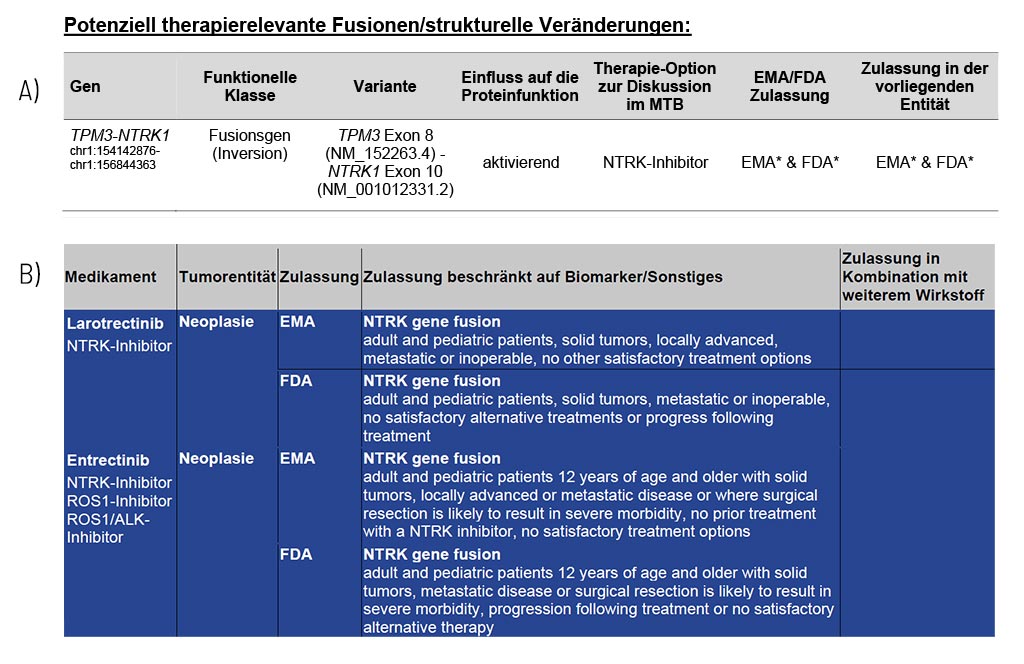
Die identifizierten Fusionen werden zusammen mit den daraus resultierenden Informationen zu den möglichen Therapieoptionen, einschließlich der EMA/FDA-Zulassung, im Befund detailliert dargestellt (A). Diese Therapieoptionen sind die Grundlage für die Spezialistinnen und Spezialisten eines molekularen Tumorboards (MTB), um eine individuelle Behandlungsempfehlung für jede Patientin und jeden Patienten zu diskutieren.
Im Anhang des medizinischen Befunds listen wir für jede identifizierte Strukturvariante relevante Therapeutika auf (B). Diese Zusammenstellung gibt Ihnen Auskunft über Medikamentenklassen und -namen, deren Zulassung (FDA/EMA) und damit verbundene Einschränkungen.
Beispielbefund: Exemplarisch für die befundete TPM3-NTRK1-Fusion und die sich daraus ergebenden therapeutischen Optionen bei einer Person mit kolorektalem Karzinom. Oberer Teil (A): Ein Ausschnitt aus Tabelle 1 des Befunds, in dem Fusionen/strukturelle Varianten mit therapeutischer Relevanz aufgeführt sind. Unterer Teil (B): Auflistung und Beschreibung der Medikamente.
Genverzeichnis
Genliste für RNA-basierte Fusionstranskriptanalyse (CancerFusionRx®, STR01)
Genliste für de-novo Fusionstranskripterkennung
ABL1, ACTB, AFAP1, AGK, AKAP4, AKAP9, AKAP12, AKT1, AKT2, AKT3, ALK, ARHGAP6, ARHGAP26, ASPL, ASPSCR1, ATF1, ATP1B1, ATRX, AVIL, AXL, BAG4, BCL2, BCOR, BCORL1, BCR, BEND2, BICC1, BRAF, BRD3, BRD4, c11orf95, CAMTA1, CCAR2, CCDC6, CCDC88A, CCDC170, CCNB3, CCND1, CD44, CD74, CEP85L, CIC, CLDN18, CLIP1, CLTC, CNTRL, COL1A1, CREB1, CREB3L1, CREB3L2, CRTC1, CTNNB1, DDIT3, DNAJB1, EGFR, EML4, EPC1, EPCAM, ERBB2, ERBB4, ERG, ESR1, ESRRA, ETV1, ETV4, ETV5, ETV6, EWSR1, EZR, FEV, FGFR1, FGFR2, FGFR3, FLI1, FN1, FOXO1, FOXO4, FOXR2, FUS, GLI1, GOPC, GPR128, HEY1, HMGA2, HTRA1, IGF1R, INSR, JAK2, JAZF1, KIAA1549, KIF5B, KIT, LEUTX, LMNA, LPP, LTK, MAGI3, MAML1, MAML2, MAML3, MAMLD1, MAP3K8, MARS1, MAST1, MAST2, MEAF6, MET, MGA, MGMT, MITF, MKL2, MN1, MSH2, MYB, MYBL1, MYC, NAB2, NCOA1, NCOA2, NCOA3, NCOA4, NFATC2, NFIB, NOTCH2, NPM1, NR4A3, NRG1, NRG2, NSD3, NTRK1, NTRK2, NTRK3, NUTM1, PAX3, PAX7, PAX8, PBX1, PDGFB, PDGFD, PDGFRA, PDGFRB, PHF1, PIK3CA, PLAG1, PML, POU5F1, PPARG, PPARGC1A, PPP1CB, PRKACA, PRKAR1A, PRKCA, PRKCB, PRKD1, PRKD2, PRKD3, PTPRZ1, QKI, RAD51B, RAF1, RANBP2, RARA, RELA, RELCH, RET, ROS1, RPS6KB1, RREB1, RSPO2, RSPO3, SDC1, SDC4, SH3PXD2A,SLC1A2, SHTN1, SLC34A2, SLC44A1, SLC45A3, SND1, SQSTM1, SS18, SSX1, SSX2, SSX4, STAT6, STRN, SUZ12, TACC1, TACC2, TACC3, TAF2N, TAF15, TCF3, TCF12, TERT, TFE3, TFEB, TFG, THADA, TMPRSS2, TPM3, TPR, TRIM24, TRIM33, TRIO, TTYH1, VGLL2, VGLL3, VMP1, WT1, WWTR1, YAP1, YWHAE, ZC3H7B, ZMYM2, ZNF703
Genliste für ausgewählte Bruchpunkte in diesen Fusiongenen
AFAP1-NTRK2, ATP1B1-NRG1, BCOR-CCNB3, BRD3-NUTM1, BRD4-NUTM1, CCDC6–RET, CCDC88A-ALK, CD74-NRG1, CD74-ROS1, CLTC-ALK, DNAJB1-PRKACA, EGFR-PPARGC1A, EML4-ALK, ETV6 NTRK2, ETV6-NTRK3, EWSR1-ATF1, EWSR1-ERG, EWSR1-FLI1, EWSR1-WT1, EZR-ROS1, FGFR1-TACC1, FGFR2-BICC1, FGFR2 TACC3, FGFR3-TACC3, KIAA1549-BRAF, KIF5B-ALK, KIF5B–RET, MGA-NUTM1, NAB2-STAT6, NCOA4–RET, NPM1-ALK, NSD3-NUTM1, PAX3-FOXO1, PAX7-FOXO1, PPP1CB-ALK, PRKAR1A-RET, QKI-NTRK2, RANBP2- ALK, RPS6KB1–VMP1, SDC4-NRG1, SDC4 ROS1, SLC34A2-ROS1, SND1-BRAF, SS18-SSX1, SS18-SSX2, STRN ALK, TMPRSS2-ERG, TPM3-ALK, TPM3-NTRK1, TPM3-ROS1, TPR NTRK1, TRIM24-BRAF, TRIM24-NTRK2, TRIM33-RET, TRIO-TERT
Liste für spezifische Transkriptvarianten
EGFR del ex2-22 (mLEEK), EGFR del ex25-26 (EGFRvIVb), EGFR del ex25-27 (EGFRvIVa), EGFR del ex26-27, EGFR del ex14-15 (vII), EGFR del ex2-7 (vIII), FGFR2IIIb, MET ex14 skipping, NFE2L2 ex2 skipping, PDGFRA del ex8-9
Downloads
(STR01 auf Seite 3 des CancerPrecision® Einsendeformular)
Kontaktieren Sie uns
Sie haben noch eine Frage oder Interesse an unserem Service?
Diagnostik-Support
Wir unterstützen Sie auf Wunsch bei der Auswahl der diagnostischen Strategie – für jede einzelne Patientin und jeden einzelnen Patienten.