Die Einführung von Next-Generation Sequencing (NGS) hat unsere Herangehensweise an wissenschaftliche und diagnostische Fragen grundlegend verändert. Das Potenzial von NGS, große Mengen an Sequenzierdaten zu einem verhältnismäßig niedrigen Preis zu erzeugen, führte zu einer Reihe bahnbrechender Entdeckungen und leitete eine neue Ära in der Genomik ein. Third-Generation Sequencing hat dank der Single-Molecule-Real-Time-Technologie (SMRT) den Nachweis von Genmutationen revolutioniert.
Die Kombination von fünf Illumina NovaSeq™ 6000 und zwei NovaSeq™ X Plus-Sequenziersystemen für Hochdurchsatzsequenzierungen, den zwei MiSeq-Seuquenzierplattformen und dem PacBio’s Sequell IIe für qualitativ hochwertige Long-Read-Sequenzierung, bietet ein breites Spektrum an Sequenzieroptionen für Forschungsprojekte und Pharmastudien.
CeGaT hat sich seit der Gründung auf den Einsatz von NGS spezialisiert. Wir investieren kontinuierlich in die neuesten Technologien, um für unsere Kundinnen und Kunden präzise und zuverlässige Ergebnisse zu erzielen. So können wir unseren Kundinnen und Kunden inzwischen sogar Third Generation Sequencing (TGS) auf der PacBio Sequenzierplattform anbieten. Unser fundiertes diagnostisches Wissen und unsere bioinformatische Expertise ergänzen die hochmodernen Laborprozesse und Sequenziertechnologien. Wir bieten das Wissen, die neueste Technologie und einen preisgekrönten Service für Ihre Projekte.
Unser Team verfügt über umfangreiche Erfahrungen mit menschlichen und anderen Säugetierproben sowie über ein breites Spektrum an Fachwissen bei der Bearbeitung von pflanzlichen und mikrobiellen Proben.
Durch den langjährigen Einsatz der NGS-Technologie haben wir viel Erfahrung in der Hochdurchsatztechnologie für die Bearbeitung tausender Proben erworben. Unsere Labore sind mit Liquid-Handling-Plattformen von Hamilton ausgestattet. Diese Plattformen können individuell programmiert werden, so dass wir unsere Protokolle gezielt an Ihre Anforderungen anpassen können.
Die Multiplexing-Strategie, die wir in unserem Labor etabliert haben, ermöglicht die gleichzeitige Sequenzierung von Hunderten von Proben in einem Sequenzierlauf oder sogar in einer Lane und stellt somit eine echte Hochdurchsatz-Sequenzierung dar.
Gerne demonstrieren wir Ihnen die Machbarkeit Ihrer Hochdurchsatzprojekte in einer Pilotstudie.
Sequenzierplattformen
Illumina NovaSeq™ X Plus System
- Für Sequenzierung mit hohem Durchsatz und außergewöhnlicher Genauigkeit
- Ideal für die Durchführung größerer Studien und Daten-intensiver Projekte
- Erzeugt pro Lauf mit zwei Flow Cells bis zu 16 Tb Daten in 48 Stunden
- Typische Anwendungen sind:
- Genomsequenzierung
- Exomsequenzierung
- Gezielte Sequenzierung
Finden Sie hier einen Performance-Vergleich unserer NovaSeq™ Geräte.

Illumina NovaSeq™ 6000 System
- Für Short-Read-Sequenzierung mit hohem Durchsatz und niedrigsten Kosten pro Base
- Ideal für Großprojekte und Deep-Sequencing-Projekte
- Erzeugt bis zu 6 Tb Daten in 20 Stunden
- Typische Anwendungen sind:
- Genomsequenzierung
- Gezielte Sequenzierung (z. B. Exomsequenzierung)
- RNA Sequenzierung
- Translationale Onkologie
- Microbiomanalyse
- Ready-to-Load-Sequenzierung
Finden Sie hier einen Performance-Vergleich unserer NovaSeq™ Geräte.

Illumina MiSeq™ System
- Für kleinere Sequenzierprojekte mit mehreren Konfigurationen
- Readlänge bis zu 2 x 300 bp
- Typische Anwendungen sind:
- Amplikon-Sequenzierung
- 16S Sequenzierung
- Sequenzierung kleiner Genome

Illumina Workflow
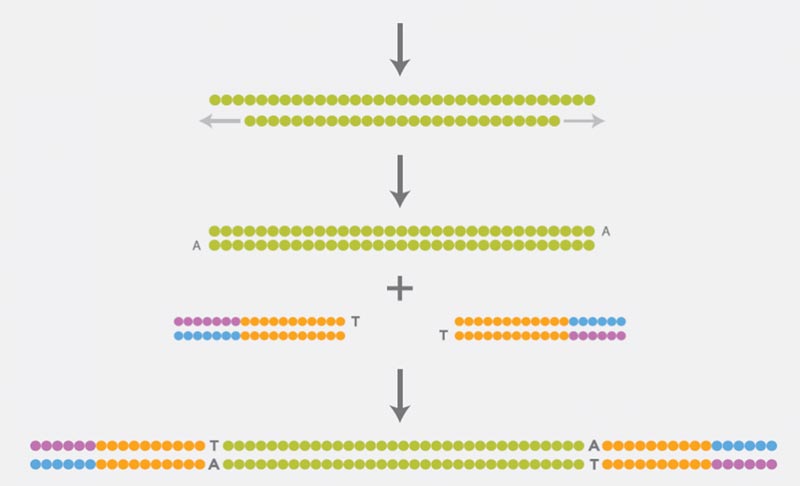
Konstruktion der Sequenzier-Library
Für die Konstruktion der Sequenzier-Library muss die DNA zunächst fragmentiert werden. Die Enden der DNA-Fragmente werden anschließend repariert, da sie durch die Fragmentierung beschädigt werden. Mit der Ligation von Adaptern an beide Enden der reparierten DNA-Fragmente ist die Herstellung der DNA-Library abgeschlossen. Diese Adapter enthalten Sequenzmotive, die für die nachfolgenden Schritte (klonale Amplifikation und die eigentliche Sequenzierung) benötigt werden.
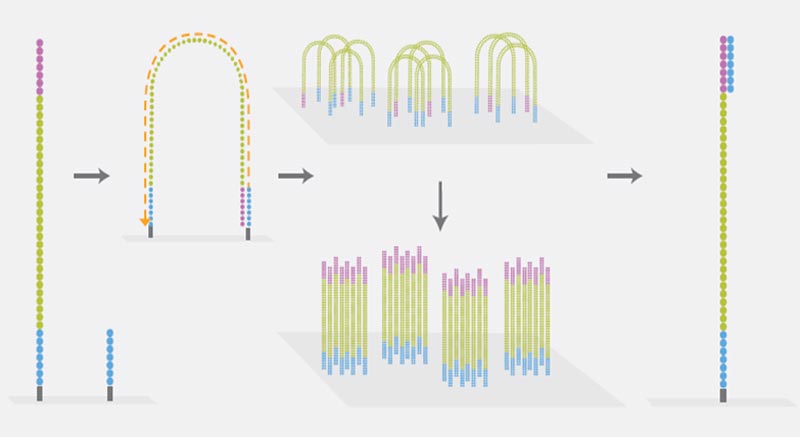
Klonale Amplifikation
Die Illumina-Sequenziertechnologie verwendet die sogenannte “Bridge-PCR” zur klonalen Amplifikation einzelner DNA-Fragmente. Diese Art der PCR wird auf der Flow Cell durchgeführt, auf der auch die eigentliche Sequenzierung stattfindet. Die DNA-Fragmente werden über die Adapter an die Flow Cell gebunden. Anschließend wird die Bridge PCR durchgeführt. Die an die Flow Cell gebundenen Oligos fungieren dabei als Primer, und jedes einzelne DNA-Fragment bildet ein separates Cluster identischer DNA-Fragmente.
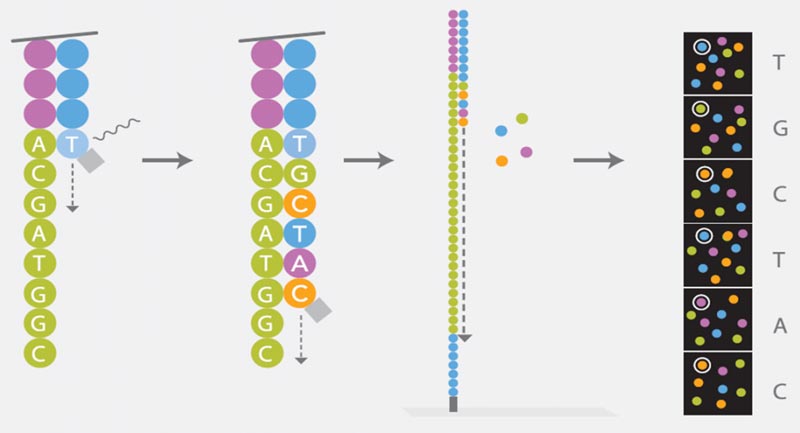
Sequenzierung
Die Sequencing-by-Synthesis-Methode (SBS, Sequenzierung durch Synthese) von Illumina basiert auf dem Prinzip der so genannten Cyclic Reversible Termination, kurz CRT. Jedes der vier Nukleotide ist mit einem anderen Farbstoff gekoppelt und ist durch eine Terminator-Gruppe modifiziert. Während eines Reaktionszyklus werden alle vier Nukleotide gleichzeitig der Polymerase zur Synthese des DNA-Strangs angeboten. Nach dem Einbau eines komplementären Nukleotids ist eine Verlängerung aufgrund der blockierenden Wirkung der Terminatorgruppe nicht mehr möglich. Die vier Farbstoffe jedes Nukleotids können mit Hilfe bildgebender Verfahren nachgewiesen werden. Der Farbstoff und die Terminatorgruppe werden abgespalten und ein neuer Synthesezyklus beginnt. Die Sequenz eines jeden Clusters wird somit gleichzeitig Base für Base bestimmt.
PacBio Sequel® IIe System
- Für HiFi-Sequenzierung von Long-Reads in hoher Qualität
- Für Sequenzierprojekte mit Reads bis zu 25 kb und einer Genauigkeit von 99,9%
- Typische Anwendungen sind:
- De novo Genomsequenzierung
- RNA Sequenzierung
- Transkriptomsequenzierung in voller Länge (Iso-Seq)
- 16S Sequenzierung in voller Länge

PacBio Workflow
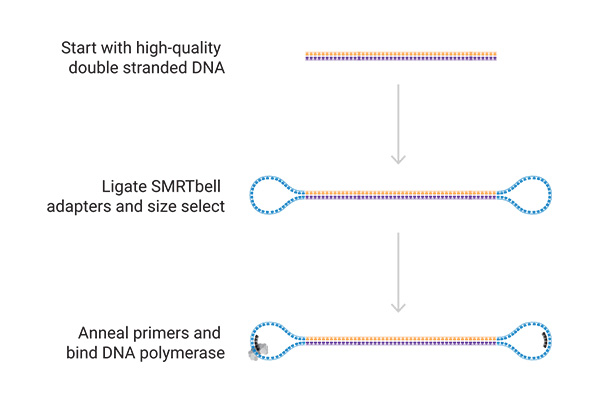
Preparation of a Library
Zunächst wird qualitativ hochwertige DNA oder RNA isoliert. Anschließend wird eine SMRTBell®-Library durch die Ligation von Adaptern an die doppelsträngige DNA erzeugt, wodurch ein ringförmiges Template entsteht. Dabei werden die DNA-Fragmente auf beiden Seiten mit ligierten Hairpin-Adaptern verschlossen. Anschließend werden die Library Primer hinzugefügt, die an die Adapter gebunden werden können. Es entsteht ein ringförmiges Template, in dem sich die Polymerase bewegt.
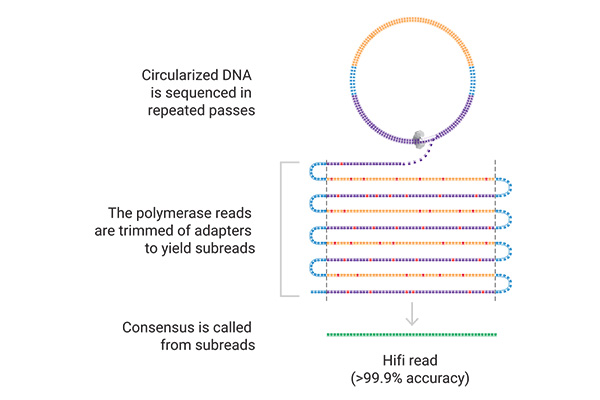
Sequenzierung
Die SMRT®-Cell enthält Millionen von Vertiefungen, so genannte Zero-Mode-Waveguides (ZMWs). Einzelne DNA-Moleküle werden in diesen Vertiefungen immobilisiert, und die Polymerase baut fluoreszenzmarkierte Nukleotide ein. Bei diesem Prozess wird Licht emittiert und der Nukleotideinbau in Echtzeit gemessen. Die Reaktionen werden aufgezeichnet und können analysiert werden. Bei der Sequenzierung im Circular Consensus Mode (CCS) werden hochpräzise Long Reads, sogenannte Hifi Reads erzeugt, da bei der SMRT-Sequenzierung dasselbe DNA-Molekül mehrfach sequenziert werden kann.
10x Genomics® Chromium™ X Controller
- Für die Analyse einzelner Zellen im großen Maßstab
- Mikrofluidik-System zur Erfassung zehntausender einzelner Zellen pro Probe
- Typische Anwendungen sind:
- Einzelzell-RNA-Sequenzierung
- Einzelzell-Immunprofiling
- Einzelzell-Epigenomik

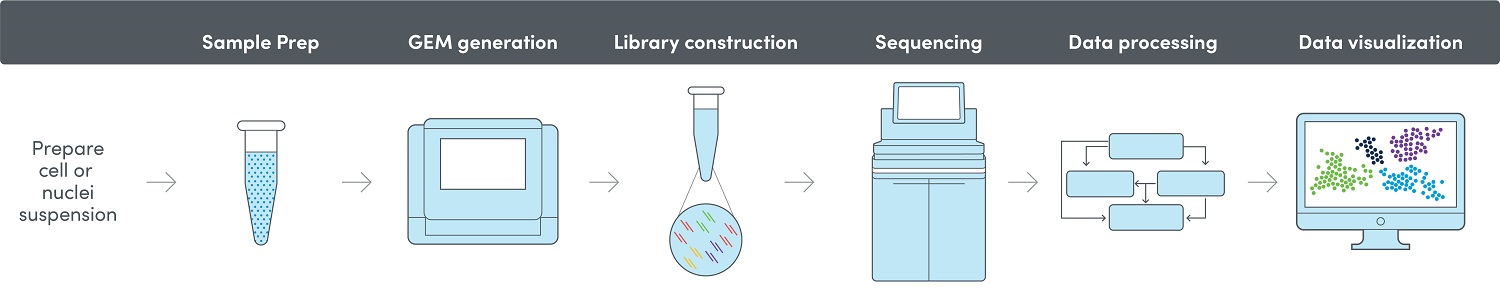
10x Genomics® Workflow
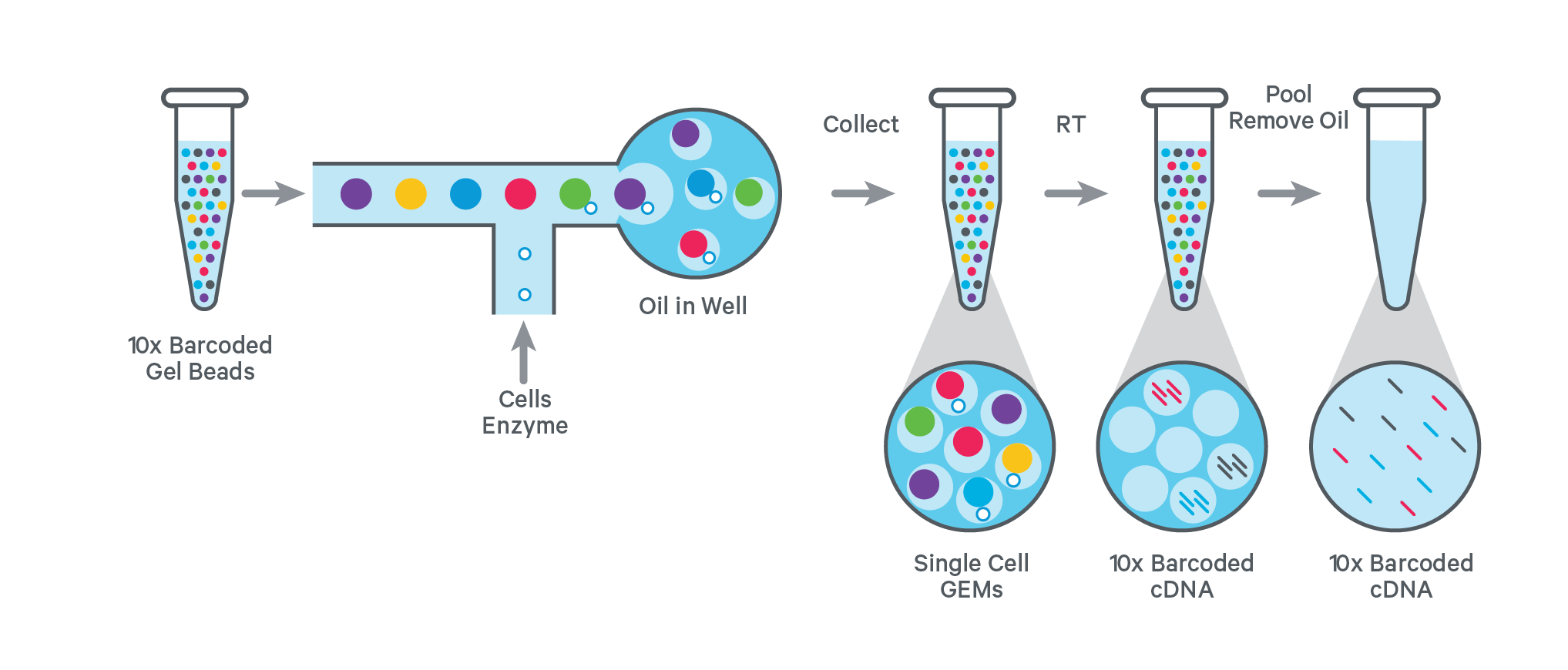
Erzeugung von Gel Beads in Emulsion
Der Arbeitsablauf zur Herstellung einer Single-Cell-Library beginnt mit der Kombination von Einzelzellsuspensionen mit einem Reagenzienmix und 10x barcodierten Gelbeads. Auf dem Chromium-Chip werden sogenannte Gel Beads in Emulsion (GEMs) erzeugt, indem Zellen und Gel Beads in einem Flüssigkeitsstrom in einem Kanal innerhalb des Chips kombiniert werden. Beim Passieren der Grenzfläche zum Öl bilden sich Tröpfchen, die gesammelt werden. Die meisten der gesammelten GEMs enthalten ein Gelkügelchen und ein Teil enthält eine einzelne Zelle.
Konstruktion der Library und Sequenzierung
Nach der GEM-Herstellung lösen sich die Gelkügelchen auf und geben Oligos und Enzyme in die Lösung ab, um die Amplifikation zu ermöglichen. Das Ergebnis sind viele Kopien einer Zelle mit demselben Barcode, aber einer eindeutigen molekularen Kennung (Unique molecular Identifier, kurz UMI). Die barcodierten Fragmente werden vereinigt (engl. Pooling), und die endgültige Konstruktion der Library wird durchgeführt.
Die Sequenzierung der Librarys wird auf dem NovaSeq™ 6000- oder MiSeq-Sequenziersystem von Illumina unter Verwendung des 10x-Systems durchgeführt.
Kontaktieren Sie uns
Sie haben noch Fragen oder Interesse an unserem Service? Treten Sie gern mit uns in Kontakt. Wir werden uns schnellstmöglich um Ihr Anliegen kümmern.
Starten Sie Ihr Projekt mit uns
Gerne beraten wir Sie zu unseren Sequenzierdienstleitungen und erarbeiten mit Ihnen gemeinsam die beste Lösung, die auf Ihre klinische Studie oder Forschungsprojekt abgestimmt ist.
Bitte geben Sie, falls möglich, folgende Probeninformationen an: Ausgangsmaterial, Anzahl der Proben, bevorzugte Option für die Vorbereitung der Library, bevorzugte Sequenziertiefe und gewünschte bioinformatische Analysestufe.